Прописи цифра один: Прописи с цифрой 1 — Kid-mama
Прописи “Математические прописи. Учимся писать цифры. 1 класс”, Узорова О.В., 25022
Прописи “Математические прописи. Учимся писать цифры. 1 класс”, Узорова О.В., 25022Онлайн гипермаркет
ЗАКАЗЫ ПРИНИМАЮТСЯ КРУГЛОСУТОЧНО
Пн-Пт с 10:00 до 18:00 СБ-ВС – выходные
0- Главная
- Книги
- Детям и родителям
- Подготовка к школе
- Прописи “Математические прописи. Учимся писать цифры. 1 класс”, Узорова О.В., 25022
Бонусные баллы: 3
690687
78. 91
91
от 5 дней
Минимальное кол-во: 2
В корзину
Все товары бренда АСТ
Работа с прописью способствует формированию навыков каллиграфического письма цифр, учит соблюдать единый орфографический режим при решении примеров и задач. Для младшего школьного возраста.
| ISBN | 978-5-17-024665-6 |
| Автор | Узорова О.В. |
| Возрастное ограничение | 6+ |
| Год издания | 2019 |
| Издательство | АСТ |
| Количество страниц | 48 |
| Объем | 0.0001 |
| Серия | Быстрое обучение |
| Страна | Россия |
| Тематика | Дополнительная литература по естественнонаучным предметам (1-4 кл) |
| Тип обложки | мягкая |
| Язык издания | русский |
Дополнительно
Личный кабинет
Обратная связь
© 2021. Канцтовары Brauberg
Канцтовары Brauberg
«Математические прописи. Учимся писать цифры. 1 класс» – описание книги | Быстрое обучение: методика О.В. Узоровой
Алматы
Алтайский край
Ангарск
Астрахань
Белгород
Благовещенск
Богучар
Братск
Брянск
Владивосток
Владимирская область
Волгоград
Волгоградская область
Воронежская область
Губкин
Екатеринбург
Ивановская область
Иркутск
Кабардино-Балкарская Республика
Калужская
Кемерово
Кемеровская область
Киров
Краснодарский край
Красноярск
Красноярский край
Курганская
Курск
ЛипецкЛиски
Москва
Московская область
Нижегородская область
Нижний Новгород
Нижний Тагил
Новосибирск
Нур-Султан
Омск
Оренбург
Оренбургская областьОрловская область
Пенза
Пермь
Республика Адыгея
Республика Башкортостан
Республика Крым
Республика Мордовия
Республика Северная Осетия — Алания
Республика Татарстан
Республика Хакасия
Россошь
Ростов-на-Дону
Ростовская область
Рязань
Самара
Самарская область
Саратов
Саратовская область
Севастополь
Смоленск
Ставрополь
Ставропольский край
Старый Оскол
Тамбов
Тамбовская область
Томск
Тула
Тулун
Тюмень
Ульяновск
Ульяновская область
Хабаровск
Ханты-Мансийский автономный округ
Челябинск
Челябинская область
Чувашская Республика
Энгельс
Ярославль
Ярославская область
Учимся писать цифры : 1-й класс : .
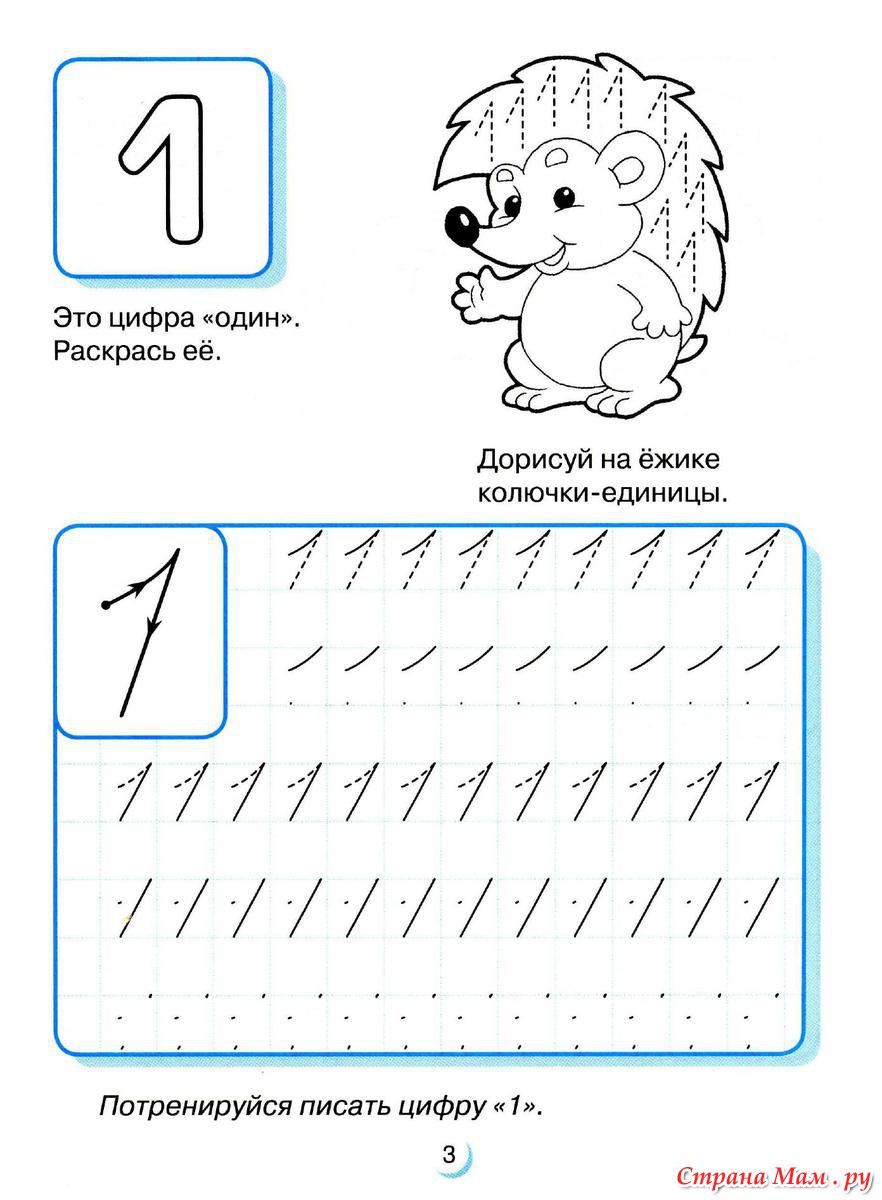 .. Узорова, О. В.
.. Узорова, О. В.Математические прописи могут использоваться для занятий с первоклассниками, при подготовке детей к школе, а также для коррекции написания цифр в начальной школе. Работа с прописью способствует формированию навыков каллиграфического письма цифр, учит соблюдать единый орфографический режим при решении примеров и задач.
Полная информация о книге
- Вид товара:Книги
- Рубрика:Математика
- Целевое назначение:Рабочие тетр.,тесты и др. уч. пособ. д/уч.1-4 кл.
- ISBN:978-5-17-024665-6
- Серия:Несерийное издание
- Издательство: АСТ
- Год издания:2020
- Тираж:10000
- Формат:70х90/16
- Штрихкод:9785170246656
- Переплет:обл.

- Сведения об ответственности:О. В. Узорова, Е. А. Нефедова
- Вес, г.:63
- Код товара:75039
Математические прописи. Учимся писать цифры. 1 класс
2кл.Математика.Вычитание с переходом через десяток.(Наглядный тренажер,АСТ)
2кл. Математика. Вычитание с переходом через десяток. (Наглядный тренажер, АСТ)
ISBN: 978-5-17-113479-2
42163
РейтингАвтор:
Наличие: Нет40 ք
2кл.Математика.Сложение с переходом через десяток.(Наглядный тренажер,АСТ)
2кл. Математика. Сложение с переходом через десяток. (Наглядный тренажер, АСТ)
ISBN: 978-5-17-113477-8
42164
РейтингАвтор:
Наличие: Нет40 ք
3кл. Математика.Внетабличное умножение в пределах 100.(Наглядный тренажер,АСТ)
Математика.Внетабличное умножение в пределах 100.(Наглядный тренажер,АСТ)
3кл. Математика. Внетабличное умножение в пределах 100. (Наглядный тренажер, АСТ)
ISBN: 978-5-17-113480-8
42165
РейтингАвтор:
Наличие: 340 ք
3кл.Математика.Умножение.Табличные случаи.(Наглядный тренажер)
3кл. Математика. Умножение. Табличные случаи. (Наглядный тренажер)
ISBN: 978-5-17-113485-3
42167
РейтингАвтор:
Наличие: Нет40 ք
1кл.Математика.Вычитание в пределах 10.(Наглядный тренажер,АСТ)
1кл. Математика. Вычитание в пределах 10. (Наглядный тренажер, АСТ)
ISBN: 978-5-17-113482-2
42159
РейтингАвтор:
Наличие: 240 ք
1кл.Математика.Сложение в пределах 10.(Наглядный тренажер,АСТ)
1кл. Математика. Сложение в пределах 10. (Наглядный тренажер, АСТ)
ISBN: 978-5-17-113483-9
42160
РейтингАвтор:
Наличие: 140 ք
2кл.Математика.Вычитание в пределах 100. (Наглядный тренажер,АСТ)
(Наглядный тренажер,АСТ)
2кл. Математика. Вычитание в пределах 100. (Наглядный тренажер, АСТ)
ISBN: 978-5-17-113481-5
42162
РейтингАвтор:
Наличие: Нет40 ք
3кл.Математика.Деление.Табличные случаи.(Наглядный тренажер,АСТ)
3кл. Математика. Деление. Табличные случаи. (Наглядный тренажер, АСТ)
ISBN: 978-5-17-113484-6
42166
РейтингАвтор:
Наличие: Нет40 ք
Учим таблицу умножения.(Круглый отличник)
Учим таблицу умножения. (Круглый отличник)
ISBN: 978-5-17-113977-3
41960
РейтингАвтор:
Наличие: Нет200 ք
4кл.3000 примеров по математике.Счет в пределах 1000.С ответами и методическими рекомендациями.
4кл. 3000 примеров по математике. Счет в пределах 1000. С ответами и методическими рекомендациями.
ISBN: 978-5-17-113650-5
41954
РейтингАвтор: Узорова
70 ք
4кл.3000 примеров по математике.Все виды примеров с ответами и методическими рекомендациями. (3000 пр
(3000 пр
4кл. 3000 примеров по математике. Все виды примеров с ответами и методическими рекомендациями. (3000 пр
ISBN: 978-5-17-113645-1
41953
РейтингАвтор: Узорова
70 ք
3кл.3000 примеров по математике.Табличное умножение.С ответами и методическими рекомендациями.(3000
3кл. 3000 примеров по математике. Табличное умножение. С ответами и методическими рекомендациями. (3000
ISBN: 978-5-17-113647-5
41952
РейтингАвтор: Узорова
70 ք
Книга “Математические прописи: Учимся писать цифры. 1 класс” из жанра Рабочие тетради, прописи, альбомы
Математические прописи: Учимся писать цифры. 1 класс
Автор: Узорова О.В.
Жанр: Рабочие тетради, прописи, альбомы
Издательство: АСТ
Год: 2014 Количество страниц: 48
Формат:
PDF (2. 40 МБ) 40 МБ) Дата загрузки: 24 июля 20152017-09-12 Скачать с нашего сайта Скачать в два клика
| |||
| Аннотация »Математические прописи» способствуют формированию навыков каллиграфического письма цифр, развитию мелкой моторики, учат соблюдать единый орфографический режим. | |||
| Комментарии
| |||
– обзор
Вариации числа копий в геноме
M. fascicularis Вариации числа копий ДНК (CNV) – это генетические различия в нормальной популяции, отображаемые как микроскопически невидимые делеции или амплификации участков геномной ДНК в диапазоне от 1 кб и до мегабазного масштаба [14]. CNV геномных сегментов обычно обнаруживаются в геномах людей [15] и модельных организмов, таких как мышь, крыса или муха Drosophila melanogaster [16].У людей к настоящему времени идентифицировано более 2,3 миллиона различных CNV, отображающих примерно 200 000 геномных областей [17]. Они вносят значительный вклад в генетическую изменчивость, охватывая большее количество нуклеотидов на геном, чем SNP (например, примерно 0,8% длины генома человека различается между двумя людьми) [18]. Поскольку CNV могут находиться в областях генома, несущих гены, очевидно, что CNV в форме гомозиготных делеций действуют как мутанты с нокаутом генов, что приводит к полной потере экспрессии и функции генов.Однако помимо этого было показано, что CNV не только влияют на экспрессию генов, различающихся по количеству копий (амплификации или гетерозиготные делеции по сравнению с эталоном), но также влияют на тканевые транскриптомы в глобальном масштабе, изменяя уровень и время экспрессии. генов, которые локализуются в CNV [16,19] и на ее флангах [20-24], эффект, который может распространяться на всю длину пораженной хромосомы [25]. Эти эффекты CNV трудно понять и не обязательно предсказуемы.Тем не менее, понимание полного влияния CNV на регуляцию экспрессии генов помогает обеспечить механистическое понимание заболеваний, связанных с CNV, либо за счет дозировки одного гена, таких как восприимчивость к СПИДу [26], гломерулонефрит у крыс и людей [27] и псориаз.
Они вносят значительный вклад в генетическую изменчивость, охватывая большее количество нуклеотидов на геном, чем SNP (например, примерно 0,8% длины генома человека различается между двумя людьми) [18]. Поскольку CNV могут находиться в областях генома, несущих гены, очевидно, что CNV в форме гомозиготных делеций действуют как мутанты с нокаутом генов, что приводит к полной потере экспрессии и функции генов.Однако помимо этого было показано, что CNV не только влияют на экспрессию генов, различающихся по количеству копий (амплификации или гетерозиготные делеции по сравнению с эталоном), но также влияют на тканевые транскриптомы в глобальном масштабе, изменяя уровень и время экспрессии. генов, которые локализуются в CNV [16,19] и на ее флангах [20-24], эффект, который может распространяться на всю длину пораженной хромосомы [25]. Эти эффекты CNV трудно понять и не обязательно предсказуемы.Тем не менее, понимание полного влияния CNV на регуляцию экспрессии генов помогает обеспечить механистическое понимание заболеваний, связанных с CNV, либо за счет дозировки одного гена, таких как восприимчивость к СПИДу [26], гломерулонефрит у крыс и людей [27] и псориаз. [28], или через изменения экспрессии непрерывного набора генов, такие как синдром Вильямса-Бёрена и детские спазмы [29,30], синдром ДиДжорджи [31], синдром Смита-Магениса [32], синдром Потоцкого-Лупски [33] ] и расстройства аутистического спектра [34–36].Более того, влияние CNV на экспрессию генов дает понимание и ассоциацию индивидуальной вариабельности фармакологических ответов, с CNV CYP2D6 в качестве наиболее яркого примера. Было показано, что такие CNV вызывают у людей невосприимчивость к терапии антидепрессантами, очевидно более высокую частоту суицидов и токсичность после лечения анальгетическим кодеином [37].
[28], или через изменения экспрессии непрерывного набора генов, такие как синдром Вильямса-Бёрена и детские спазмы [29,30], синдром ДиДжорджи [31], синдром Смита-Магениса [32], синдром Потоцкого-Лупски [33] ] и расстройства аутистического спектра [34–36].Более того, влияние CNV на экспрессию генов дает понимание и ассоциацию индивидуальной вариабельности фармакологических ответов, с CNV CYP2D6 в качестве наиболее яркого примера. Было показано, что такие CNV вызывают у людей невосприимчивость к терапии антидепрессантами, очевидно более высокую частоту суицидов и токсичность после лечения анальгетическим кодеином [37].
Учитывая важность CNV в биомедицинских исследованиях, мы разработали первый полногеномный тайлинг-массив яванских макак, содержащий 4.2 миллиона 60-мерных зондов, которые опрашивают весь геном в среднем с интервалом в 600 пар оснований. Из сканирования генома исключены повторяющиеся области центромер и Y-хромосома, которые еще не секвенированы. В качестве эталона CNV мы использовали объединенную ДНК, выделенную от 25 самцов яванского макака маврикийского происхождения. Использование мужской ДНК в качестве базовой имеет то преимущество, что женские хромосомы можно использовать в качестве эталона для дупликаций генов и производительности массива. На рисунке 4.1 мы показываем пример плотности тайлинг-зонда в области 60 т.п.н. на хромосоме 1 с усилением CNV и области в 90 т.п.н. на хромосоме 19 с сигналом потери CNV, оба у самца макаки из Маврикия. .Амплификация влияет на интрон гена кальпонина 3 ( CNN3 ), необходимый для слияния трофобластических клеток [38], тогда как делеция содержит весь локус, кодирующий кристаллический галектин Шарко-Лейдена ( CLC ). По аналогии с CNV для интронных регуляторных элементов трансмембранного регулятора проводимости при муковисцидозе, амплифицированный сегмент может влиять на сплайсинг транскриптов и, следовательно, на экспрессию генов [39]. Потеря CNV в этом примере, скорее всего, повлияет на уровни экспрессии гена CLC , который связан с предрасположенностью к аллергическому риниту [40].
Использование мужской ДНК в качестве базовой имеет то преимущество, что женские хромосомы можно использовать в качестве эталона для дупликаций генов и производительности массива. На рисунке 4.1 мы показываем пример плотности тайлинг-зонда в области 60 т.п.н. на хромосоме 1 с усилением CNV и области в 90 т.п.н. на хромосоме 19 с сигналом потери CNV, оба у самца макаки из Маврикия. .Амплификация влияет на интрон гена кальпонина 3 ( CNN3 ), необходимый для слияния трофобластических клеток [38], тогда как делеция содержит весь локус, кодирующий кристаллический галектин Шарко-Лейдена ( CLC ). По аналогии с CNV для интронных регуляторных элементов трансмембранного регулятора проводимости при муковисцидозе, амплифицированный сегмент может влиять на сплайсинг транскриптов и, следовательно, на экспрессию генов [39]. Потеря CNV в этом примере, скорее всего, повлияет на уровни экспрессии гена CLC , который связан с предрасположенностью к аллергическому риниту [40].
Рисунок 4. 1. Обнаружение вариаций числа копий (CNV) у одной яванской макаки с Маврикия. Область размером 60 т.п.н. на хромосоме 1 и область размером 90 т.п.н. на хромосоме 19 были выбраны в качестве примеров для обнаружения CNV у одной маврикийской яванской макаки (вверху). Схематические изображения местоположений генов и экзонов в этих геномных областях показаны в середине вместе с геномными положениями тайлинговых зондов. Зазоры, не покрытые датчиками, либо повторяются, либо не уникальны, а высота стержня на датчик представляет собой увеличение потенциальной неоднозначности датчика.Сигналы CNV по сравнению с эталонным стандартом отображаются как отношение log2 (черные точки), показывая усиление CNV (синий) и потери (красный), обнаруженные алгоритмом SegMNT (красные линии).
1. Обнаружение вариаций числа копий (CNV) у одной яванской макаки с Маврикия. Область размером 60 т.п.н. на хромосоме 1 и область размером 90 т.п.н. на хромосоме 19 были выбраны в качестве примеров для обнаружения CNV у одной маврикийской яванской макаки (вверху). Схематические изображения местоположений генов и экзонов в этих геномных областях показаны в середине вместе с геномными положениями тайлинговых зондов. Зазоры, не покрытые датчиками, либо повторяются, либо не уникальны, а высота стержня на датчик представляет собой увеличение потенциальной неоднозначности датчика.Сигналы CNV по сравнению с эталонным стандартом отображаются как отношение log2 (черные точки), показывая усиление CNV (синий) и потери (красный), обнаруженные алгоритмом SegMNT (красные линии).
В качестве следующего шага мы исследовали CNV среди группы отдельных макак cynomolgus. Мы выбрали трех самок, происходящих из китайского центра разведения (Юго-Восточная Азия) и Маврикия. Основываясь на представлении всех точек данных на хромосоме с низким разрешением, мы указали, в качестве примера, две области CNV на хромосомах 4 и 13, которые показывают значительную потерю CNV, и одну область CNV на хромосоме 7, показывающую прирост CNV у всех людей (рис. 4.2). В том же масштабе мы обнаружили выигрыш или потерю CNV, которые происходят только у одного из лиц, которые отмечены красными треугольниками на рис. 4.2. Мы расширили этот анализ CNV на весь геном на 24 обезьяны cynomolgus и определили, что среднее количество CNV на геном составляет около 800, при средней длине около 12,5 т.п.н. на локус CNV. Эти числа вполне сопоставимы с наблюдениями, сделанными для CNV в нормальных человеческих популяциях, где большинство CNV имеют небольшие размеры (<10-30 т.п.н.) [17,41] и где любой человек несет в среднем около 670 сегментов CNV [41].Кроме того, мы всесторонне проанализировали частоту областей CNV у этих 24 обезьян и обнаружили «частные» и общие CNV (рис. 4.3). В связи с этим мы обнаружили, что CNV яванского макака неслучайно распределены по геному, что указывает на концентрацию в хромосомных «горячих точках», где CNVs, вероятно, будут возникать либо de novo, либо на протяжении всей эволюции. Это открытие предполагает строгие селективные механизмы, лежащие в основе генеза или поддержания CNV.
4.2). В том же масштабе мы обнаружили выигрыш или потерю CNV, которые происходят только у одного из лиц, которые отмечены красными треугольниками на рис. 4.2. Мы расширили этот анализ CNV на весь геном на 24 обезьяны cynomolgus и определили, что среднее количество CNV на геном составляет около 800, при средней длине около 12,5 т.п.н. на локус CNV. Эти числа вполне сопоставимы с наблюдениями, сделанными для CNV в нормальных человеческих популяциях, где большинство CNV имеют небольшие размеры (<10-30 т.п.н.) [17,41] и где любой человек несет в среднем около 670 сегментов CNV [41].Кроме того, мы всесторонне проанализировали частоту областей CNV у этих 24 обезьян и обнаружили «частные» и общие CNV (рис. 4.3). В связи с этим мы обнаружили, что CNV яванского макака неслучайно распределены по геному, что указывает на концентрацию в хромосомных «горячих точках», где CNVs, вероятно, будут возникать либо de novo, либо на протяжении всей эволюции. Это открытие предполагает строгие селективные механизмы, лежащие в основе генеза или поддержания CNV.
Рисунок 4.2. Полногеномное отображение профилей CNV у cynomolgus обезьян.Синие стрелки обозначают сохраненные приросты или потери CNV, присутствующие у всех трех человек. Красные треугольники обозначают «частные» CNV. Для каждой обезьяны указано географическое происхождение. Обратите внимание на сдвиг Х-хромосомы в правом конце дисплея в результате сравнения трех самок обезьян с эталонным эталоном самцов.
Рисунок 4.3. Идеограммы участков CNV у яванских макак. Идеограммы показывают области CNV и частоты CNV для 24 обезьян cynomolgus при разрешении хромосом. Обычные CNV отражаются длинными столбцами, а «частные» CNV – короткими столбцами.Прибыли CNV отмечены синим цветом, а убытки – красным.
Затем мы задали вопрос, группируются ли профили CNV в соответствии с географическим происхождением приматов. Основываясь на относительно небольшом наборе SNP, Street et al. обнаружили, что по крайней мере некоторые из этих вариаций действительно ограничиваются географическим происхождением обезьян [42]. Более актуальным для биомедицинских исследований является тот факт, что половина этих SNP являются общими с макаками-резусами [42]. Это подразумевает, что «частные» SNP яванского макака полезны для обнаружения геномной интеграции резус-последовательностей в результате межвидовой гибридизации.Эти наблюдения делают вероятным, что CNV как индикаторы генетической рекомбинации также будут группироваться в соответствии с географическим происхождением, что и имело место (рис. 4.4). Основные аутосомные профили CNV 22 из 24 обезьян cynomolgus группируются в зависимости от происхождения (Филиппины, Юго-Восточная Азия или Маврикий). Это снова означает, что хромосомные целевые локусы для CNV неслучайно распределены по всему геному и что многие локусы CNV «фиксированы» в пределах географической субпопуляции и внутри семейств макак cynomolgus, что согласуется с данными SNP.Если теперь принять во внимание, что скорость генерации SNP de novo составляет около 69 новых SNP на геном потомства у людей [43] и что скорость генерации de novo человеческих CNV составляет около 1,2 новых CNV на 100 геномов потомства [44], тогда мы можем предположить, что скорость образования CNV de novo у яванских макак довольно низка.
Более актуальным для биомедицинских исследований является тот факт, что половина этих SNP являются общими с макаками-резусами [42]. Это подразумевает, что «частные» SNP яванского макака полезны для обнаружения геномной интеграции резус-последовательностей в результате межвидовой гибридизации.Эти наблюдения делают вероятным, что CNV как индикаторы генетической рекомбинации также будут группироваться в соответствии с географическим происхождением, что и имело место (рис. 4.4). Основные аутосомные профили CNV 22 из 24 обезьян cynomolgus группируются в зависимости от происхождения (Филиппины, Юго-Восточная Азия или Маврикий). Это снова означает, что хромосомные целевые локусы для CNV неслучайно распределены по всему геному и что многие локусы CNV «фиксированы» в пределах географической субпопуляции и внутри семейств макак cynomolgus, что согласуется с данными SNP.Если теперь принять во внимание, что скорость генерации SNP de novo составляет около 69 новых SNP на геном потомства у людей [43] и что скорость генерации de novo человеческих CNV составляет около 1,2 новых CNV на 100 геномов потомства [44], тогда мы можем предположить, что скорость образования CNV de novo у яванских макак довольно низка. Это, в свою очередь, привело нас к выводу, что генетическая адаптация может управлять поддержанием количества копий в этих конкретных локусах. Первоначальный случай адаптивных аллелей CNV при положительном отборе был недавно обнаружен у людей при изменении дозировки гена альфа-амилазы 1A ( AMY1A ).Этот ген кодирует белок, который катализирует первый этап переваривания диетического крахмала и гликогена. Число копий AMY1A у людей в три раза выше, чем у шимпанзе, что позволяет предположить, что людям отдается предпочтение в дозировке гена из-за сопутствующего увеличения потребления крахмала [45]. Таким образом, вероятно, что генетическая адаптация отдельных CNV яванского макака может влиять на уровни экспрессии генов, обеспечивая селективное преимущество для условий окружающей среды, таких как питание.Мы проверили эту гипотезу, проанализировав локус размером 2 Mb на хромосоме 19, где мы обнаружили делецию размером примерно 13 kb у двух яванских макак из Юго-Восточной Азии, которая затрагивает межгенный регион на этой хромосоме.
Это, в свою очередь, привело нас к выводу, что генетическая адаптация может управлять поддержанием количества копий в этих конкретных локусах. Первоначальный случай адаптивных аллелей CNV при положительном отборе был недавно обнаружен у людей при изменении дозировки гена альфа-амилазы 1A ( AMY1A ).Этот ген кодирует белок, который катализирует первый этап переваривания диетического крахмала и гликогена. Число копий AMY1A у людей в три раза выше, чем у шимпанзе, что позволяет предположить, что людям отдается предпочтение в дозировке гена из-за сопутствующего увеличения потребления крахмала [45]. Таким образом, вероятно, что генетическая адаптация отдельных CNV яванского макака может влиять на уровни экспрессии генов, обеспечивая селективное преимущество для условий окружающей среды, таких как питание.Мы проверили эту гипотезу, проанализировав локус размером 2 Mb на хромосоме 19, где мы обнаружили делецию размером примерно 13 kb у двух яванских макак из Юго-Восточной Азии, которая затрагивает межгенный регион на этой хромосоме. У нас были данные микроматрицы экспрессии генов, доступные для сердца, почек, печени, легких и селезенки шести животных дикого типа для этого локуса в дополнение к двум животным с потерей числа копий. Неожиданно потеря числа копий межгенного сегмента хромосомы статистически значимо модулировала уровни экспрессии соседних генов в почках и сердце, но не в других тканях.Например, уровни мРНК активаторов транскрипции FOXA3, MEIS3, NPAS1 и SIX5 были увеличены в почках и сердце, тогда как убиквитинлигаза IRF2BP1, SUMO-активирующий фермент SAE1, каркасный белок STRN4, переносчик аминокислот SLC1A5. , белок p190RhoGAP (GRLF1), активирующий Rho-GTPase, и регулятор слияния везикул NAPA были подавлены по сравнению с здоровыми животными (рис. 4.5). Достаточно интересно то, что делеция влияет на экспрессию примерно 45 генов, которые кодируются на расстоянии до 1 МБ выше и ниже этого CNV (данные не показаны).Более того, кажется, что существует тенденция к понижающей модуляции генов ближе к потере числа копий, тогда как более далекие гены, по-видимому, активируются.
У нас были данные микроматрицы экспрессии генов, доступные для сердца, почек, печени, легких и селезенки шести животных дикого типа для этого локуса в дополнение к двум животным с потерей числа копий. Неожиданно потеря числа копий межгенного сегмента хромосомы статистически значимо модулировала уровни экспрессии соседних генов в почках и сердце, но не в других тканях.Например, уровни мРНК активаторов транскрипции FOXA3, MEIS3, NPAS1 и SIX5 были увеличены в почках и сердце, тогда как убиквитинлигаза IRF2BP1, SUMO-активирующий фермент SAE1, каркасный белок STRN4, переносчик аминокислот SLC1A5. , белок p190RhoGAP (GRLF1), активирующий Rho-GTPase, и регулятор слияния везикул NAPA были подавлены по сравнению с здоровыми животными (рис. 4.5). Достаточно интересно то, что делеция влияет на экспрессию примерно 45 генов, которые кодируются на расстоянии до 1 МБ выше и ниже этого CNV (данные не показаны).Более того, кажется, что существует тенденция к понижающей модуляции генов ближе к потере числа копий, тогда как более далекие гены, по-видимому, активируются. Уровни экспрессии генов за пределами этого локуса размером 2 Mb демонстрируют относительно высокую индивидуальную вариабельность, что затрудняет интерпретацию данных. На данный момент мы можем только предполагать механизм, лежащий в основе влияния удаленного участка ДНК на всю хромосому. Такие механизмы могут включать физическую диссоциацию активаторов или репрессоров транскрипции, изменение структуры хроматина или изменение положения геномной области в ядре.
Уровни экспрессии генов за пределами этого локуса размером 2 Mb демонстрируют относительно высокую индивидуальную вариабельность, что затрудняет интерпретацию данных. На данный момент мы можем только предполагать механизм, лежащий в основе влияния удаленного участка ДНК на всю хромосому. Такие механизмы могут включать физическую диссоциацию активаторов или репрессоров транскрипции, изменение структуры хроматина или изменение положения геномной области в ядре.
Рисунок 4.4. Географическая кластеризация по профилям CNV. Дендрограммы показывают взаимосвязь кластеров CNV, обнаруженных в аутосомах 24 отдельных обезьян cynomolgus. Пол обозначен буквами M для мужчин и F для женщин, а цветные полосы указывают на происхождение (красный = Юго-Восточная Азия; синий = Маврикий; зеленый = Филиппины).
Рисунок 4.5. Связь между CNV и экспрессией генов. Показана область размером 2 Mb на хромосоме 19, содержащая более 45 локусов генов. Две из восьми обезьян cynomolgus несут делецию размером примерно 13 килобаз в центре этой геномной области. Изменение экспрессии генов показано для генов, на которые сильно влияет CNV в носителе, по сравнению с обезьянами, не являющимися носителями, на основании данных микроматрицы для сердца и почек. Красные стрелки указывают на активацию экспрессии генов, а синие стрелки – на отрицательную. Коды отдельных обезьян cynomolgus появляются на оси и .
Изменение экспрессии генов показано для генов, на которые сильно влияет CNV в носителе, по сравнению с обезьянами, не являющимися носителями, на основании данных микроматрицы для сердца и почек. Красные стрелки указывают на активацию экспрессии генов, а синие стрелки – на отрицательную. Коды отдельных обезьян cynomolgus появляются на оси и .
CNV = Номер копии Варианты = Копировать изменение номера
| ||
Варианты числа копий и рак | Геномная медицина
Феук Л., Карсон А.Р., Шерер С.В.: Структурные вариации в геноме человека. Nat Rev Genet. 2006, 7: 85-97. 10.1038 / nrg1767
CAS Статья PubMed Google Scholar
Gault J, Hopkins J, Berger R, Drebing C, Logel J, Walton C, Short M, Vianzon R, Olincy A, Ross RG, Adler LE, Freedman R, Leonard S: Сравнение полиморфизмов в гене никотинового рецептора альфа7 и его частичное дублирование у шизофреников и контрольных субъектов. Am J Med Genet B Neuropsychiatr Genet. 2003, 123В: 39-49. 10.1002 / ajmg.b.20061
Статья PubMed Google Scholar
Traherne JA: Архитектура и эволюция MHC человека: значение для исследований ассоциации болезней.Int J Immunogenet. 2008, 35: 179-192. 10.1111 / j.1744-313X.2008.00765.x
PubMed Central CAS Статья PubMed Google Scholar
Яфрат А.Дж., Феук Л., Ривера М.Н., Листевник М.Л., Донахью П.К., Ци Й., Шерер С.В., Ли К. Обнаружение крупномасштабных вариаций в геноме человека. Нат Жене. 2004, 36: 949-951. 10.1038 / ng1416
CAS Статья PubMed Google Scholar
Себат Дж., Лакшми Б., Троге Дж., Александр Дж., Янг Дж., Лундин П., Манер С., Масса Х, Уокер М., Чи М., Навин Н., Лучито Р., Хили Дж., Хикс Дж., Йе К., Райнер А., Гиллиам TC, Траск Б., Паттерсон Н., Зеттерберг А., Виглер М.: Крупномасштабный полиморфизм числа копий в геноме человека. Наука. 2004, 305: 525-528. 10.1126 / science.1098918
CAS Статья PubMed Google Scholar
Stranger BE, Forrest MS, Dunning M, Ingle CE, Beazley C, Thorne N, Redon R, Bird CP, de Grassi A, Lee C, Tyler-Smith C, Carter N, Scherer SW, Tavare S , Deloukas P, Hurles ME, Dermitzakis ET: Относительное влияние вариации числа нуклеотидов и копий на фенотипы экспрессии генов.Наука. 2007, 315: 848-853. 10.1126 / science.1136678
PubMed Central CAS Статья PubMed Google Scholar
Henrichsen CN, Vinckenbosch N, Zollner S, Chaignat E, Pradervand S, Schutz F, Ruedi M, Kaessmann H, Reymond A: Сегментарные вариации количества копий формируют транскриптомы тканей. Нат Жене. 2009, 41: 424-429. 10.1038 / ng.345
CAS Статья PubMed Google Scholar
Гурьев В., Саар К., Адамович Т., Ферхеул М., ван Хиш С.А., Кук С., Правенец М., Эйтман Т., Джейкоб Х., Шулл Д.Д., Хубнер Н., Куппен Э.: Распределение и функциональное влияние изменения числа копий ДНК у крысы . Нат Жене. 2008, 40: 538-545. 10.1038 / ng.141
CAS Статья PubMed Google Scholar
Иноуэ К., Лупски Дж. Р.: Молекулярные механизмы геномных нарушений. Анну Рев Геномикс Хум Генет. 2002, 3: 199-242. 10.1146 / аннурьев.геном.3.032802.120023
CAS Статья PubMed Google Scholar
Прасад С.Е., Хоули С., Мерфи К.С.: Гены-кандидаты и поведенческий фенотип при синдроме делеции 22q11.2. Dev Disabil Res Rev.2008, 14: 26-34. 10.1002 / ddrr.5
Артикул PubMed Google Scholar
Биттел Д.К., Батлер М.Г.: Синдром Прадера-Вилли: клиническая генетика, цитогенетика и молекулярная биология.Эксперт Rev Mol Med. 2005, 7: 1-20. 10.1017 / S1462399405009531
Артикул PubMed Google Scholar
Мейер-Линденберг A, Мервис CB, Берман KF: Нейронные механизмы при синдроме Вильямса: уникальное окно для генетических влияний на познание и поведение. Nat Rev Neurosci. 2006, 7: 380-393. 10.1038 / nrn1906
CAS Статья PubMed Google Scholar
Elsea SH, Girirajan S: синдром Смита-Магениса. Eur J Hum Genet. 2008, 16: 412-421. 10.1038 / sj.ejhg.5202009
CAS Статья PubMed Google Scholar
Merla G, Howald C, Henrichsen CN, Lyle R, Wyss C, Zabot MT, Antonarakis SE, Reymond A: Субмикроскопическая делеция у пациентов с синдромом Вильямса-Бойрена влияет на уровни экспрессии негемизиготных фланкирующих генов. Am J Hum Genet. 2006, 79: 332-341. 10.1086/506371
PubMed Central CAS Статья PubMed Google Scholar
Клейньян Д.А., ван Хейнинген V: Контроль экспрессии генов на больших расстояниях: новые механизмы и нарушения в болезни. Am J Hum Genet. 2005, 76: 8-32. 10.1086 / 426833
PubMed Central CAS Статья PubMed Google Scholar
Тернер Д.Д., Миретти М., Раджан Д., Фиглер Х., Картер Н.П., Блейни М.Л., Бек С., Херлс М.Э .: Частота мейотических делеций и дупликаций de novo de novo в зародышевой линии, вызывающих несколько геномных нарушений.Нат Жене. 2008, 40: 90-95. 10.1038 / ng.2007.40
PubMed Central CAS Статья PubMed Google Scholar
Махер Б. Личные геномы: случай отсутствия наследственности. Природа. 2008, 456: 18-21. 10.1038 / 456018a
CAS Статья PubMed Google Scholar
Shlien A, Tabori U, Marshall CR, Pienkowska M, Feuk L, Novokmet A, Nanda S, Druker H, Scherer SW, Malkin D: Чрезмерная вариация числа копий геномной ДНК при синдроме предрасположенности к раку Ли-Фраумени .Proc Natl Acad Sci USA. 2008, 105: 11264-11269. 10.1073 / pnas.0802970105
PubMed Central CAS Статья PubMed Google Scholar
Хиггинс М.Э., Клермонт М., Майор Дж. Э., Сандер С., Лэш А. Е.: Рак-гены: ресурс по селекции генов для проектов генома рака. Nucleic Acids Res. 2007, 35: D721-726. 10.1093 / nar / gkl811
PubMed Central CAS Статья PubMed Google Scholar
Redon R, Ishikawa S, Fitch KR, Feuk L, Perry GH, Andrews TD, Fiegler H, Shapero MH, Carson AR, Chen W, Cho EK, Dallaire S, Freeman JL, Gonzalez JR, Gratacos M, Huang J, Kalaitzopoulos D, Komura D, MacDonald JR, Marshall CR, Mei R, Montgomery L, Nishimura K, Okamura K, Shen F, Somerville MJ, Tchinda J, Valsesia A, Woodwark C, Yang F: Глобальные вариации числа копий в геноме человека . Природа. 2006, 444: 444-454. 10.1038 / nature05329
PubMed Central CAS Статья PubMed Google Scholar
МакКэрролл С.А., Курувилла Ф.Г., Корн Дж. М., Коули С., Немеш Дж., Вайсокер А., Шаперо М. Х., де Баккер П. И., Маллер Дж. Б., Кирби А., Эллиотт А. Л., Паркин М., Хаббелл Е., Вебстер Т., Мей Р., Вейтч Дж., Коллинз П.Дж., Хэндсакер Р., Линкольн С., Низзари М., Блюм Дж., Джонс К.В., Рава Р., Дейли М.Дж., Габриэль С.Б., Альтшулер Д.: Комплексное обнаружение и популяционно-генетический анализ SNP и вариации числа копий. Нат Жене. 2008, 40: 1166-1174. 10.1038 / ng.238
CAS Статья PubMed Google Scholar
Forbes SA, Bhamra G, Bamford S, Dawson E, Kok C, Clements J, Menzies A, Teague JW, Futreal PA, Stratton MR: Каталог соматических мутаций при раке (COSMIC). Curr Protoc Hum Genet. 2008, Глава 10 (Блок 10): 11-
PubMed Google Scholar
Thomas G, Jacobs KB, Kraft P, Yeager M, Wacholder S, Cox DG, Hankinson SE, Hutchinson A, Wang Z, Yu K, Chatterjee N, Garcia-Closas M, Gonzalez-Bosquet J, Prokunina -Olsson L, Orr N, Willett WC, Colditz GA, Ziegler RG, Berg CD, Buys SS, McCarty CA, Feigelson HS, Calle EE, Thun MJ, Diver R, Prentice R, Jackson R, Kooperberg C, Chlebowski R, Lissowska Дж .: Многоступенчатое исследование ассоциации генома при раке груди выявило два новых аллеля риска в 1p11.2 и 14q24.1 (RAD51L1). Нат Жене. 2009, 41: 579-584. 10.1038 / ng.353
PubMed Central CAS Статья PubMed Google Scholar
Перри Г.Х., Бен-Дор А., Цаленко А., Сампас Н., Родригес-Ревенга Л., Тран С.В., Шеффер А., Штайнфельд И., Цанг П, Ямада Н.А., Парк Х.С., Ким Джи, Сео Дж.С., Яхини З., Ладерман С., Брун Л., Ли К.: Мелкомасштабная и сложная архитектура изменения числа копий человека. Am J Hum Genet. 2008, 82: 685-695.10.1016 / j.ajhg.2007.12.010
PubMed Central CAS Статья PubMed Google Scholar
Франк Б., Бермеджо Дж. Л., Хемминки К., Саттер С., Ваппеншмидт Б., Майндл А., Кихле-Бахат М., Бугерт П., Шмутцлер Р. К., Бартрам С. Р., Бурвинкель В. Вариант числа копий в гене-кандидате, подавляющем опухоль. MTUS1 и риск семейного рака груди. Канцерогенез. 2007, 28: 1442-1445. 10.1093 / carcin / bgm033
CAS Статья PubMed Google Scholar
МакКэрролл С.А., Альтшулер Д.М.: исследования вариаций числа копий и ассоциации болезней человека. Нат Жене. 2007, 39: S37-S42. 10.1038 / ng2080
CAS Статья PubMed Google Scholar
Ionita-Laza I, Rogers AJ, Lange C, Raby BA, Lee C: Анализ генетической ассоциации вариации числа копий (CNV) в патогенезе болезней человека. Геномика. 2009, 93: 22-26. 10.1016 / j.ygeno.2008.08.012
PubMed Central CAS Статья PubMed Google Scholar
Надь Р., Sweet K, Eng C: синдромы наследственного рака с высокой проникающей способностью. Онкоген. 2004, 23: 6445-6470. 10.1038 / sj.onc.1207714
CAS Статья PubMed Google Scholar
Schouten JP, McElgunn CJ, Waaijer R, Zwijnenburg D, Diepvens F, Pals G: относительное количественное определение 40 последовательностей нуклеиновых кислот с помощью мультиплексной амплификации зонда, зависимой от лигирования. Nucleic Acids Res. 2002, 30: e57- 10.1093 / nar / gnf056
PubMed Central Статья PubMed Google Scholar
Futreal PA, Coin L, Marshall M, Down T, Hubbard T, Wooster R, Rahman N, Stratton MR: Перепись генов рака человека. Nat Rev Рак. 2004, 4: 177-183. 10.1038 / nrc1299
PubMed Central CAS Статья PubMed Google Scholar
Jackson EM, Shaikh TH, Gururangan S, Jones MC, Malkin D, Nikkel SM, Zuppan CW, Wainwright LM, Zhang F, Biegel JA: анализ массива однонуклеотидных полиморфизмов высокой плотности у пациентов с делециями зародышевой линии 22q11.2 и злокачественная рабдоидная опухоль. Hum Genet. 2007, 122: 117-127. 10.1007 / s00439-007-0386-3
CAS Статья PubMed Google Scholar
Ханахан Д., Вайнберг Р.А.: Признаки рака. Клетка. 2000, 7: 57-70. 10.1016 / S0092-8674 (00) 81683-9. 10.1016 / S0092-8674 (00) 81683-9
Артикул Google Scholar
Weir BA, Woo MS, Getz G, Perner S, Ding L, Beroukhim R, Lin WM, Province MA, Kraja A, Johnson LA, Shah K, Sato M, Thomas RK, Barletta JA, Borecki IB , Broderick S, Chang AC, Chiang DY, Chirieac LR, Cho J, Fujii Y, Gazdar AF, Giordano T, Greulich H, Hanna M, Johnson BE, Kris MG, Lash A, Lin L, Lindeman N: характеристика генома рака при аденокарциноме легкого.Природа. 2007, 450: 893-898. 10.1038 / nature06358
PubMed Central CAS Статья PubMed Google Scholar
Маллиган К.Г., Гурха С., Радтке И., Миллер С.Б., Кустан-Смит Э, Далтон Дж. Д., Гиртман К., Мэтью С., Ма Дж., Фунт С.Б., Су Х, Пуй СН, Реллинг М.В., Эванс В.Е., Шуртлефф С.А., Даунинг Дж. Р.: Полногеномный анализ генетических изменений при остром лимфобластном лейкозе. Природа. 2007, 446: 758-764. 10.1038 / nature05690
CAS Статья PubMed Google Scholar
,: Комплексная геномная характеристика определяет гены глиобластомы человека и основные пути. Природа. 2008, 455: 1061-1068. 10.1038 / nature07385
Артикул Google Scholar
Hodgson SV, Coonar AS, Hanson PJ, Cottrell S, Scriven PN, Jones T., Hawley PR, Wilkinson ML: Два случая делеций 5q у пациентов с семейным аденоматозным полипозом: возможная связь с болезнью Кароли. J Med Genet. 1993, 30: 369-375. 10.1136 / jmg.30.5.369
PubMed Central CAS Статья PubMed Google Scholar
Su LK, Steinbach G, Sawyer JC, Hindi M, Ward PA, Lynch PM: Геномные перестройки гена-супрессора опухоли APC при семейном аденоматозном полипозе. Hum Genet. 2000, 106: 101-107. 10.1007 / s0043016
CAS Статья PubMed Google Scholar
Aretz S, Stienen D, Uhlhaas S, Pagenstecher C, Mangold E, Caspari R, Propping P, Friedl W. Большие субмикроскопические делеции геномных APC являются частой причиной типичного семейного аденоматозного полипоза.J Med Genet. 2005, 42: 185-192. 10.1136 / jmg.2004.022822
PubMed Central CAS Статья PubMed Google Scholar
Charames GS, Ramyar L, Mitri A, Berk T, Cheng H, Jung J, Bocangel P, Chodirker B, Greenberg C, Spriggs E, Bapat B: большая новая делеция в промоторной области APC вызывает ген молчание и приводит к классическому семейному аденоматозному полипозу у родственников меннонитов из Манитобы. Hum Genet. 2008, 124: 535-541.10.1007 / s00439-008-0579-4
CAS Статья PubMed Google Scholar
Delnatte C, Sanlaville D, Mougenot JF, Vermeesch JR, Houdayer C, Blois MC, Genevieve D, Goulet O, Fryns JP, Jaubert F, Vekemans M, Lyonnet S, Romana S, Eng C, Stoppa- Lyonnet D: Непрерывная делеция гена в плече 10q хромосомы связана с ювенильным полипозом в младенчестве, что отражает кооперацию между генами-супрессорами опухолей BMPR1A и PTEN.Am J Hum Genet. 2006, 78: 1066-1074. 10.1086 / 504301
PubMed Central CAS Статья PubMed Google Scholar
Petrij-Bosch A, Peelen T., van Vliet M, van Eijk R, Olmer R, Drusedau M, Hogervorst FB, Hageman S, Arts PJ, Ligtenberg MJ, Meijers-Heijboer H, Klijn JG, Vasen HF , Cornelisse CJ, van ‘t Veer LJ, Bakker E, van Ommen GJ, Devilee P: геномные делеции BRCA1 являются основными мутациями-основателями у голландских пациентов с раком груди.Нат Жене. 1997, 17: 341-345. 10.1038 / ng1197-341
CAS Статья PubMed Google Scholar
Montagna M, Dalla Palma M, Menin C, Agata S, De Nicolo A, Chieco-Bianchi L, D’Andrea E: геномные перестройки составляют более одной трети мутаций BRCA1 в груди северной Италии. / Семьи с раком яичников. Hum Mol Genet. 2003, 12: 1055-1061. 10.1093 / hmg / ddg120
CAS Статья PubMed Google Scholar
Casilli F, Tournier I, Sinilnikova OM, Coulet F, Soubrier F, Houdayer C, Hardouin A, Berthet P, Sobol H, Bourdon V, Muller D, Fricker JP, Capoulade-Metay C, Chompret A, Nogues C, Mazoyer S , Chappuis P, Maillet P, Philippe C, Lortholary A, Gesta P, Bezieau S, Toulas C, Gladieff L, Maugard CM, Provencher DM, Dugast C, Delvincourt C, Nguyen TD, Faivre L: Вклад перестройки зародышевой линии в спектр мутаций BRCA2. J Med Genet. 2006, 43: e49-10.1136 / jmg.2005.040212
PubMed Central CAS Статья PubMed Google Scholar
Lesueur F, de Lichy M, Barrois M, Durand G, Bombled J, Avril MF, Chompret A, Boitier F, Lenoir GM, Bressac-de Paillerets B, Baccard M, Bachollet B, Berthet P, Bonadona V, Bonnetblanc JM, Карон О, Шеврант-Бретон Дж., Куни Дж. Ф., Далле С., Делоне М., Деманж Л., Де Катребарб Дж., Доре Дж. Ф., Френей М., Фрикер Дж. П., Готье-Виллар М., Геста П., Жиро С., Горри П., Гранж Ф: Вклад крупных геномных делеций в локусе CDKN2A в бремя семейной меланомы. Br J Рак. 2008, 99: 364-370.10.1038 / sj.bjc.6604470
PubMed Central CAS Статья PubMed Google Scholar
Cybulski C, Wokolorczyk D, Huzarski T, Byrski T, Gronwald J, Gorski B, Debniak T, Masojc B, Jakubowska A, Gliniewicz B, Sikorski A, Stawicka M, Antczakias Z , Krajka K, Lauer W, Sosnowski M, Sikorska-Radek P, Bar K, Klijer R, Zdrojowy R, Malkiewicz B, Borkowski A, Borkowski T, Szwiec M, Narod SA, Lubinski J: большая делеция зародышевой линии в киназе Chek2 ген связан с повышенным риском рака простаты.J Med Genet. 2006, 43: 863-866. 10.1136 / jmg.2006.044974
PubMed Central CAS Статья PubMed Google Scholar
Cybulski C, Wokolorczyk D, Huzarski T, Byrski T., Gronwald J, Gorski B, Debniak T., Masojc B, Jakubowska A, Wetering van de T, Narod SA, Lubinski J: удаление A в CHEK2 из 5 , 395 п.н. предрасполагают к раку груди в Польше. Лечение рака груди Res. 2007, 102: 119-122. 10.1007 / s10549-006-9320-у
CAS Статья PubMed Google Scholar
Левран О., Диотти Р., Пуджара К., Батиш С.Д., Ханенберг Х., Ауэрбах А.Д.: Спектр вариаций последовательности в гене FANCA: исследование Международного реестра анемии Фанкони (IFAR). Hum Mutat. 2005, 25: 142-149. 10.1002 / humu.20125
CAS Статья PubMed Google Scholar
van Hattem WA, Brosens LA, де Ленг WW, Morsink FH, Lens S, Carvalho R, Giardiello FM, Offerhaus GJ: Большие геномные делеции SMAD4, BMPR1A и PTEN при ювенильном полипозе.Кишечник. 2008, 57: 623-627. 10.1136 / gut.2007.142927
CAS Статья PubMed Google Scholar
Киши М., Цукада Т., Симидзу С., Футами Х., Ито Y, Канбе М., Обара Т., Ямагути К.: большая делеция гена MEN1 в зародышевой линии в семье с множественной эндокринной неоплазией 1 типа. Jpn J Cancer Res. 1998, 89: 1-5.
CAS Статья PubMed Google Scholar
Nystrom-Lahti M, Kristo P, Nicolaides NC, Chang SY, Aaltonen LA, Moisio AL, Jarvinen HJ, Mecklin JP, Kinzler KW, Vogelstein B: Основополагающие мутации и Alu-опосредованная рекомбинация при наследственном раке толстой кишки. Nat Med. 1995, 1: 1203-1206. 10,1038 / нм 1195-1203
CAS Статья PubMed Google Scholar
Chan TL, Yuen ST, Ho JW, Chan AS, Kwan K, Chung LP, Lam PW, Tse CW, Leung SY: новая делеция hMLh2 размером 1,8 kb в зародышевой линии, имитирующая альтернативный сплайсинг: мутация-основатель в китайское население.Онкоген. 2001, 20: 2976-2981. 10.1038 / sj.onc.1204376
CAS Статья PubMed Google Scholar
Stella A, Surdo NC, Lastella P, Barana D, Oliani C, Tibiletti MG, Viel A, Natale C, Piepoli A, Marra G, Guanti G: новые делеции MSh3 зародышевой линии и делеция MSh3-основателя, связанная с эффекты ожидания в HNPCC. Clin Genet. 2007, 71: 130-139. 10.1111 / j.1399-0004.2007.00745.x
CAS Статья PubMed Google Scholar
Plaschke J, Ruschoff J, Schackert HK: Геномные перестройки hMSH6 способствуют генетической предрасположенности при подозрении на наследственный синдром неполипозного колоректального рака. J Med Genet. 2003, 40: 597-600. 10.1136 / jmg.40.8.597
PubMed Central CAS Статья PubMed Google Scholar
Riva P, Corrado L, Natacci F, Castorina P, Wu BL, Schneider GH, Clementi M, Tenconi R, Korf BR, Larizza L: синдром микроделеции NF1: уточненная FISH-характеристика спорадических и семейных делеций с локусом -специфические зонды.Am J Hum Genet. 2000, 66: 100-109. 10.1086 / 302709
PubMed Central CAS Статья PubMed Google Scholar
Бауш Б., Бороздин В., Нойманн HP: Клинические и генетические характеристики пациентов с нейрофиброматозом 1 типа и феохромоцитомой. N Engl J Med. 2006, 354: 2729-2731. 10.1056 / NEJMc066006
CAS Статья PubMed Google Scholar
Цилчорозиду Т., Менко Ф.Х., Лаллоо Ф., Кидд А., Де Силва Р., Томас Х., Смит П., Малкольмсон А., Доре Дж., Мадан К., Браун А., Йовос Дж. Г., Цалигопулос М., Вогиатзис Н., Басер М. Э., Уоллес А. Дж., Эванс Д.Г.: Конституционные перестройки хромосомы 22 как причина нейрофиброматоза 2. J. Med Genet. 2004, 41: 529-534. 10.1136 / jmg.2003.016774
PubMed Central CAS Статья PubMed Google Scholar
Хорват А., Босис I, Гиацакис С., Левин Е., Вайнберг Ф., Меоли Е., Робинсон-Уайт А., Сигель Дж., Сони П., Грассен Л., Матяхина Л., Верма С., Реммерс Е., Нестерова М., Carney JA, Bertherat J, Stratakis CA: Большие делеции гена PRKAR1A в комплексе Карни.Clin Cancer Res. 2008, 14: 388-395. 10.1158 / 1078-0432.CCR-07-1155
CAS Статья PubMed Google Scholar
Шимкетс Р., Гайлани М.Р., Сиу В.М., Янг-Фенг Т., Прессман С.Л., Леванат С., Голдштейн А., Дин М., Бейл А.Е.: Молекулярный анализ делеций хромосомы 9q у двух пациентов с синдромом Горлина. Am J Hum Genet. 1996, 59: 417-422.
PubMed Central CAS PubMed Google Scholar
Bremner R, Du DC, Connolly-Wilson MJ, Bridge P, Ahmad KF, Mostachfi H, Rushlow D, Dunn JM, Gallie BL: Удаление экзонов 24 и 25 RB вызывает ретинобластому с низкой пенетрантностью. Am J Hum Genet. 1997, 61: 556-570. 10.1086 / 515499
PubMed Central CAS Статья PubMed Google Scholar
Cascon A, Montero-Conde C, Ruiz-Llorente S, Mercadillo F, Leton R, Rodriguez-Antona C, Martinez-Delgado B, Delgado M, Diez A, Rovira A, Diaz JA, Robledo M: Грубые делеции SDHB у пациентов с параганглиомой, обнаруженные с помощью мультиплексной ПЦР: возможная горячая точка ?.Гены Хромосомы Рак. 2006, 45: 213-219. 10.1002 / gcc.20283
CAS Статья PubMed Google Scholar
Baysal BE, Willett-Brozick JE, Filho PA, Lawrence EC, Myers EN, Ferrell RE: Alu-опосредованная частичная делеция SDHC вызывает семейную и спорадическую параганглиому. J Med Genet. 2004, 41: 703-709. 10.1136 / jmg.2004.019224
PubMed Central CAS Статья PubMed Google Scholar
McWhinney SR, Pilarski RT, Forrester SR, Schneider MC, Sarquis MM, Dias EP, Eng C: Большие делеции зародышевой линии субъединиц митохондриального комплекса II SDHB и SDHD в наследственной параганглиоме. J Clin Endocrinol Metab. 2004, 89: 5694-5699. 10.1210 / jc.2004-0769
CAS Статья PubMed Google Scholar
Свенсен Дж., Кейзер Дж., Коффин С.М., Бигель Дж. А., Вискочил Д.Х., Уильямс М.С.: Семейное происхождение шванном и злокачественной рабдоидной опухоли, связанной с дупликацией в SMARCB1.J Med Genet. 2009, 46: 68-72. 10.1136 / jmg.2008.060152
PubMed Central CAS Статья PubMed Google Scholar
Le Meur N, Martin C, Saugier-Veber P, Joly G, Lemoine F, Moirot H, Rossi A, Bachy B, Cabot A, Joly P, Frebourg T: полная делеция зародышевой линии гена STK11 в семья с синдромом Пейтца-Егерса. Eur J Hum Genet. 2004, 12: 415-418. 10.1038 / sj.ejhg.5201155
CAS Статья PubMed Google Scholar
Bougeard G, Brugieres L, Chompret A, Gesta P, Charbonnier F, Valent A, Martin C, Raux G, Feunteun J, Bressac-de Paillerets B, Frebourg T: скрининг перегруппировок TP53 в семьях с синдромом Ли-Фраумени. полная делеция гена TP53. Онкоген. 2003, 22: 840-846. 10.1038 / sj.onc.1206155
CAS Статья PubMed Google Scholar
Bougeard G, Sesboue R, Baert-Desurmont S, Vasseur S, Martin C, Tinat J, Brugieres L, Chompret A, de Paillerets BB, Stoppa-Lyonnet D, Bonaiti-Pellie C, Frebourg T: Molecular основа синдрома Ли-Фраумени: обновленная информация из французских семей LFS.J Med Genet. 2008, 45: 535-538. 10.1136 / jmg.2008.057570
CAS Статья PubMed Google Scholar
Kozlowski P, Roberts P, Dabora S, Franz D, Bissler J, Northrup H, Au KS, Lazarus R, Domanska-Pakiela D, Kotulska K, Jozwiak S, Kwiatkowski DJ: Идентификация 54 больших делеций / дупликации в TSC1 и TSC2 с использованием MLPA и корреляции генотип-фенотип. Hum Genet. 2007, 121: 389-400. 10.1007 / s00439-006-0308-9
CAS Статья PubMed Google Scholar
Richards FM, Phipps ME, Latif F, Yao M, Crossey PA, Foster K, Linehan WM, Affara NA, Lerman MI, Zbar B: Картирование гена-супрессора опухоли болезни фон Хиппеля-Линдау: идентификация делеций зародышевой линии с помощью импульсного полевого геля электрофорез. Hum Mol Genet. 1993, 2: 879-882. 10,1093 / hmg / 2,7,879
CAS Статья PubMed Google Scholar
Huff V, Miwa H, Haber DA, Call KM, Housman D, Strong LC, Saunders GF: Доказательства того, что WT1 является геном опухоли Вильмса (WT): внутригенная делеция зародыша в двустороннем WT.Am J Hum Genet. 1991, 48: 997-1003.
PubMed Central CAS PubMed Google Scholar
Секвенирование всего генома выявляет высокую сложность вариаций числа копий в локусах устойчивости к инсектицидам у малярийных комаров
- Эрик Р. Лукас1,
- Алистер Майлз2,3,
- Николас Дж.Harding3,
- Крис С. Кларксон2,
- Мара К.Н. Lawniczak2,
- Доминик П. Квятковски2,3,
- Дэвид Уитман1,
- Мартин Дж. Доннелли1,2,
- The Anopheles gambiae Консорциум 1000 Genomes4
- 1 Ливерпульская школа тропической медицины, Ливерпуль L3 5QA, Соединенное Королевство;
- 2 Wellcome Sanger Institute, Hinxton, Cambridge CB10 1SA, United Kingdom;
- 3 Институт больших данных, Оксфордский университет, Центр медицинской информации и открытий Ли Ка Шинга, Оксфорд, OX3 7LF, United Королевство
Аннотация
Полиморфизмы в количестве генетических копий могут влиять на экспрессию генов, кодирующую последовательность и зиготность, делая их мощными участниками в эволюционном процессе. Однако варианты числа копий (CNV) недостаточно изучены, и их труднее обнаружить, чем однонуклеотидные. полиморфизмы. Мы используем преимущества интенсивного селективного давления на основной переносчик малярии Anopheles gambiae , вызванное широким использованием инсектицидов для борьбы с малярией, для исследования роли CNV в эволюции инсектицидов. сопротивление.Используя данные полногеномного секвенирования из 1142 образцов в An. gambiae 1000 геномов, мы идентифицировали 250 генов, содержащих CNV, включающих в общей сложности 267 генов, 28 из которых были в генах семьи, связанные с метаболической устойчивостью к инсектицидам, представляющие собой значительное обогащение этих семей. Пять основных все кластеры генов метаболической устойчивости содержат CNV, причем в этих кластерах обнаружено 44 различных CNV, а также несколько CNVs часто покрывают одни и те же гены.Эти 44 CNV широко распространены (45% людей носят хотя бы один из них) и распространяются посредством положительного отбора, на что указывают их высокие локальные частоты и расширенная гомозиготность гаплотипов. Наши результаты демонстрируют важность CNV в ответ на селекцию, подчеркивая срочную необходимость идентификации вклад каждого CNV в устойчивость к инсектицидам и отслеживание их распространения по мере использования инсектицидов при эндемичной малярии стран активизируется, и по мере того, как оперативное развертывание надкроватных сеток следующего поколения, направленных на борьбу с метаболической резистентностью, набирает обороты шаг.Наши подробные описания CNVs, обнаруженных во всем ареале видов, предоставляют инструменты для этого.
Варианты числа копий (CNV) – это форма генетической вариации, которая возникает, когда геномная последовательность удаляется или дублируется, потенциально влияет как на структуру, так и на уровни экспрессии кодирующих последовательностей и играет решающую роль в эволюции и адаптации (Редон и др., 2006; Чен и др., 2008; Эмерсон и др.2008 г.). Отчасти важность CNV заключается в широком диапазоне эффектов, которые они могут оказывать на транскриптом. Увеличивается количество копий (амплификации), охватывающих всю последовательность гена, может привести к повышенным уровням экспрессии, если новый ген копии связаны с цис- -регуляторными последовательностями, необходимыми для транскрипции (Handsaker et al. 2015). Альтернативно, дупликация или делеция только части последовательности гена может привести к серьезным изменениям в последовательности белка.Например, у людей CNV, охватывающий части двух генов гликофорина, создает новый гибридный гликофорин, связанный с устойчивостью. малярии (Leffler et al., 2017). CNVs также могут позволить альтернативным вариантам гена появляться в тандеме на одной хромосоме через гетерогенный ген. дупликация, создавая конститутивные гетерозиготы. Это можно увидеть у комаров Anopheles gambiae и Culex pipiens, , где мутации ацетилхолинэстеразы 1 ( Ace1 ) вызывают устойчивость к карбаматным и органофосфатным инсектицидам, но в отсутствие инсектицидов несут вред для здоровья.Эта стоимость снижается у гетерозигот, что приводит к распространению гетерогенных дупликаций Ace1 , в которых одновременно встречаются мутантные аллели и аллели дикого типа (Labbé et al. 2007; Assogba et al. 2015).
Хотя важность CNV широко признана, им обычно уделяется меньше внимания, чем однонуклеотидным полиморфизмам. (SNP) в исследованиях генетической изменчивости, вероятно, потому, что их труднее идентифицировать.Анализ генома на популяционном уровне CNVs, таким образом, редки, и степень их влияния на эволюцию плохо изучена (хотя всемирное исследование было проведено на людях [Sudmant et al. 2015]).
Малярийный комар An. gambiae и его близкий родственный вид An. coluzzii – прекрасные организмы для изучения эволюции CNV на популяционном уровне по трем причинам. Во-первых, эти виды являются основными переносчиками малярии в странах Африки к югу от Сахары (SSA) и обладают высокой антропофильностью (Kiszewski et al.2004 г.). Следовательно, они подвергаются сильной атаке со стороны инсектицидов, используемых в программах борьбы с малярией, что создает интенсивное давление отбора. это движет быстрой современной эволюцией. Таким образом, CNV имеют возможность вносить свой вклад в избирательную реакцию на это давление, обеспечение контекста, в котором можно оценить их важность. Во-вторых, CNV могут играть ключевую роль в эволюции инсектицидов. устойчивость за счет их способности влиять на экспрессию генов и допускать коэкспрессию аллелей дикого типа и мутантных аллелей (Bass and Field 2011; Weetman et al.2018). Несмотря на почти два десятилетия генетических исследований устойчивости к инсектицидам, известные SNP, связанные с устойчивостью, все еще остаются в силе. не в состоянии объяснить большую часть различий в устойчивости к инсектицидам (Donnelly et al., 2016). CNV потенциально представляют собой решающий источник недостающих вариаций, которые потенциально могут заполнить этот пробел. В-третьих, в рамках проекта Anopheles gambiae 1000 Genomes Project (Ag1000G) были получены данные полногеномного секвенирования от 1142 особей An. gambiae и An.coluzzii из нескольких мест в SSA, предоставляя уникальную возможность проводить полногеномный поиск CNV для всех видов. дистрибутивы (https://www.malariagen.net/data/ag1000g-phase-2-ar1).
Двумя основными механизмами устойчивости к инсектицидам являются устойчивость к участкам-мишеням и метаболическая устойчивость (Liu 2015). Было обнаружено, что CNV влияют на все три основных гена-мишени инсектицидов у насекомых: Ace1 (Assogba et al.2015; Weetman et al. 2015), потенциалзависимый натриевый канал para ( Vgsc ) (Martins et al. 2013, 2017) и рецептор гамма-аминомасляной кислоты (GABA) Rdl (Remnant et al. 2013), обычно сочетающий резистентные и аллели дикого типа для обеспечения устойчивости при снижении ее стоимости. Однако дублирование в Ace1 являются единственными CNV, которые, как было показано, играют роль в устойчивости к инсектицидам Anopheles , будучи связаны с мутацией, придающей устойчивость, и либо увеличивая устойчивость, это дает (Assogba et al.2016) или уменьшение стоимости пригодности мутации (Assogba et al. 2015).
Метаболические гены, уровни экспрессии которых связаны с устойчивостью к инсектицидам, были зарегистрированы у широкого круга видов. (Li et al. 2007) и вызывают особую озабоченность, поскольку активация одного гена может обеспечить перекрестную устойчивость к ряду инсектицидов. с разными целями. Например, в An. gambiae , повышающая регуляция цитохрома P450s Cyp6m2 и Cyp6p3 связана с устойчивостью к пиретроидам и бендиокарбу (Edi et al.2014). Несмотря на важность метаболической устойчивости к инсектицидам, причинные генетические варианты часто остаются неидентифицированными, хотя целенаправленные исследования выявили случаи, в которых CNV играют решающую роль в метаболической устойчивости у ряда видов. В Drosophila , дупликация гена детоксикации Cyp6g1 участвует в устойчивости к ДДТ (Schmidt et al. 2010), тогда как в Cx. quinquefasciatus , резистентность к перметрину связана с повышенной экспрессией Cyp9m10 , частично из-за дупликации (Itokawa et al.2011). Точно так же амплификация генов эстеразы, приводящая к повышенной экспрессии, обеспечивает повышенную устойчивость к органофосфатам. в комарах Сх. pipiens (Raymond et al. 2001) и Aedes albopictus (Grigoraki et al. 2015), а также к нескольким инсектицидам против персиково-картофельной тли Myzus persicae (Devonshire et al. 1998; Field et al. 1999). У коричневых цикад в результате неофункционализации дублированной копии Cyp6er1 даже был создан новый вариант гена, обеспечивающий устойчивость к неоникотиноидному имидаклоприду (Zimmer et al.2018). В ан. gambiae и An. coluzzii, наиболее важных метаболических генов, которые были идентифицированы в качестве основных кандидатов на устойчивость к инсектицидам на сегодняшний день: Gste2 (Mitchell et al. 2014), Cyp6p3 (Müller et al. 2008; Edi et al. 2014), Cyp6m2 (Стивенсон и др., 2011; Митчелл и др., 2012; Эди и др., 2014), Cyp6z1, (Чиу и др., 2008) и Cyp9k1, (Майн и др., 2015; Вонтас и др., 2018). Если CNV играют важную роль в развитии устойчивости к инсектицидам у An.gambiae и An. coluzzii , мы ожидаем найти их среди таких генов.
Здесь мы выполняем сканирование CNVs в геноме An. gambiae и An. coluzzii с использованием полногеномного секвенирования фазы 2 Ag1000G, который состоит из 1142 пойманных в дикой природе комаров, собранных из 16 популяции между 2000 и 2012 годами, секвенированные со средним охватом 32 ×. Затем мы сосредоточимся на пяти регионах, представляющих особый интерес. для метаболической устойчивости к инсектицидам, чтобы выяснить, находятся ли CNV в этих регионах под положительным отбором.
Результаты
Открытие CNV в масштабе всего генома
Сначала мы использовали данные короткочитываемого полногеномного секвенирования из набора данных Ag1000G Phase 2, чтобы найти области генома с увеличенным считайте охват относительно среднего геномного, учитывая богатство GC и исключая 27 отдельных комаров с необычно высокая дисперсия покрытия.Вкратце, мы отфильтровали геномные окна размером 300 п.н. для определения качества картирования и редких уровней содержания GC (что предотвращение нормализации) и идентифицированные области, покрывающие не менее пяти смежных окон размером 300 п.н., увеличенные не менее чем в 1,5 раза покрытие определяется скрытой марковской моделью (HMM). Затем мы отфильтровали этот исходный набор CNV на основе относительного правдоподобия наблюдаемого охвата считыванием в отсутствие CNV. После группировки идентичных CNV между людьми в соответствии с их расположение генома и сохранение только CNV, присутствующих как минимум у 5% людей в популяции (или как минимум у трех особей в небольших популяциях) мы идентифицировали 1557 кандидатов в CNV (рис.1; Дополнительные данные S1). Самым большим из них (203 100 п.н.) была хорошо задокументированная дупликация в целевом сайте инсектицида Ace1 . CNVs не были обнаружены на других распространенных участках-мишенях для инсектицидов Vgsc или Rdl .
Фигура 1.Распределение CNV в различных диапазонах размеров по геному. Синие столбцы представляют области генома, в которых CNV был найден.Числа на слева указывают диапазон размера CNV. Числа справа показывают количество CNV в этом диапазоне размеров. Моделирование показало значительное обогащение CNV в гетерохроматических областях. (0 из 10 000 имитаций имели такое же количество CNV в гетерохроматине).
Чтобы оценить точность метода обнаружения CNV, мы провели моделирование, в котором значения охвата, полученные на каждом Окно размером 300 п.н. перемешивали случайным образом.Это моделирование показало, что низкая частота CNV является результатом случайной случайности (ложное коэффициент обнаружения 0,003 на уровне выборки; 0,013 на уровне популяции) и хорошие показатели восстановления для CNV, охватывающих минимум 10 окон (≥85% на уровне выборки; ≥83% после фильтрации на уровне популяции) с более низкой скоростью восстановления для CNV охватывающий всего пять окон (31,4% на индивидуальном уровне; 42,2% после фильтрации на уровне популяции).
Чтобы определить геномные факторы, связанные с присутствием CNV, мы исследовали, как CNV распределены относительно тип хроматина и содержание генов. Мы обнаружили, что CNV были особенно распространены в гетерохроматических областях. Из 1557 CNV, 534 (34,3%) были обнаружены в гетерохроматине, который покрывает только 9,3% генома ( P <0,0002; из 10 000 симуляций количество гетерохроматических CNV варьировалось от 44 до 112, в среднем 77).CNV в гетерохроматина также были значительно больше, чем у эухроматина (медиана для гетерохроматина: 7200 п.н., медиана для эухроматина: 3300 п.н .; Тест Вилкоксона, n 1 = 534, n 2 = 1023, W = 377190, P <0,0001) (дополнительный рисунок S1).
CNV также были более многочисленны в ген-содержащих регионах. Из 1557 CNV 250 содержали по крайней мере один ген, что значительно больше, чем ожидалось случайно ( P <0.0002; через 10 000 симуляций количество CNV, содержащих гены, варьировалось от 107 до 187, в среднем 145). Поскольку эухроматин обычно содержит больше генов, чем гетерохроматин, мы повторили моделирование, сосредоточив внимание на эухроматине. CNVs и рандомизация их положения в эухроматине. Из 1023 эухроматических CNV 226 содержали по крайней мере один ген, снова представляет собой значительное обогащение генных областей ( P <0.0002; через 10 000 симуляций количество CNV, содержащих гены, варьировалось от 59 до 127, в среднем 92). Этот обогащение было постоянным для всех размеров эухроматических CNV (дополнительная таблица S1).
CNV обогащены семействами генов метаболической устойчивости
Затем мы исследовали, были ли CNV обогащены генами, потенциально участвующими в метаболической устойчивости к инсектицидам.В целом, из из 10939 генов, включенных в анализ, 267 (2,4%) находились в пределах по крайней мере одного CNV (дополнительные данные S2). Из этих 267 генов 28 были кандидатами в гены метаболической устойчивости (определяемые как цитохром P450, глутатион-S-трансфераза или карбоксилэстераза, и далее называемые «генами метаболической детоксикации»). Поскольку многие родственные гены встречаются в кластерах, и поэтому не включены в события CNV независимо друг от друга, мы подсчитали количество CNV, которые включали, по крайней мере, один метаболический детокс-ген.Из 250 CNV, содержащих какие-либо гены, 27 содержали по крайней мере один ген метаболической детоксикации, что значительно больше, чем ожидается случайно ( P <0,0002; из 10 000 симуляций количество генов CNV, содержащих гены детоксикации, варьировалось от 0 до 14, с среднее значение 4). Тот же результат был получен, когда учитывались только эухроматические CNV для всех размеров CNV (дополнительная таблица S2). Хотя между популяциями наблюдались некоторые различия в количестве генов метаболической детоксикации, обнаруженных в CNV (таблица 1), это не было значимым (точный критерий Фишера: P = 0.08).
Таблица 1.Количество обнаруженных CNV, содержащих или не содержащих хотя бы один цитохром P450, глутатион-S-трансферазу или карбоксилэстеразу (детокс-гены)
Гены, обнаруженные в CNV, были обогащены 13 терминами онтологии генов молекулярной функции (GO) после многократной корректировки до порогового значения Q , равного 0.05 (Supplemental Data S3), в первую очередь отражая обогащение двух классов генов: цитохрома P450s (важные термины GO включали монооксигеназу активность, связывание гема, связывание ионов железа, активность оксидоредуктазы) и протеазы (важные термины ГО включали несколько форм активности пептидазы). Этот результат также сохраняется, когда гетерохроматические CNV были исключены, с оставшимися 12 из 13 членов GO. значительный (дополнительные данные S3).Никакие GO-термины из онтологий биологических процессов или клеточных компартментов существенно не обогатились.
28 генов метаболической детоксикации, обнаруженных внутри CNV, были преимущественно из кластеров генов, которые ранее были вовлечены при устойчивости к инсектицидам (дополнительные данные S2), причем 16 из 28 генов являются членами эпсилон-кластера глутатион-S-трансферазы ( Gste на хромосоме 3R) или одного из четырех кластеров цитохрома P450 ( Cyp6p на хромосоме 2R, Cyp6m на хромосоме 3R, Cyp6z на хромосоме 3R и Cyp9k1 на хромосоме X).Это еще раз указывает на то, что гены, участвующие в метаболической устойчивости к инсектицидам, были в центре внимания амплификации. События. Поэтому мы выполнили подробный анализ CNV вокруг этих пяти кластеров генов (рис. 2A). Поскольку гены Cyp6aa1 / Cyp6aa2 , которые соседствуют с кластером Cyp6p , также были широко представлены в списке амплифицированных генов (дополнительные данные S2), мы расширили область исследования до Cyp6p3 , включив эти гены.
Чтобы более точно определить количество CNV в каждом из этих пяти кластеров генов, мы использовали несогласованные пары чтения и чтения. выравнивание по контрольным точкам CNV, чтобы различать разные CNV. Три из пяти кластеров генов показали большое количество различных CNVs. Мы идентифицировали 16 аллелей CNV в Cyp9k1 (названных Cyp9k1_Dup1–16) (дополнительные данные S7; дополнительный рис. S4), 15 в кластере Cyp6aa1 – Cyp6p2 (Cyp6aap_Dup1–15) (рис.2B; Дополнительные данные S4; Дополнительный рис. S1), 11 в кластере Gstu4 – Gste3 (Gstue_Dup1–11) (рис. 2C; дополнительные данные S5; дополнительный рис. S2), один на Cyp6m2 (Cyp6m_Dup1) (дополнительные данные S6) (дополнительные данные S6). Дополнительный рис. S3) и один в Cyp6z3 – Cyp6z1 (Cyp6z_Dup1) (дополнительные данные S6; дополнительный рис. S3).
Фигура 2.( A ) CNV в кластерах генов, которые, как известно, связаны с метаболической устойчивостью к инсектицидам, были обнаружены на всех трех хромосомах.( B ) Из 15 CNV в Cyp6aa1 – Cyp6p2 , 13 включают Cyp6aa1 и пять включают Cyp6p3 . На вставке карты показаны страны, в которых не менее 5% людей имели CNV в Cyp6aa1 (синий) и Cyp6p3 (красный), а страны, отсутствующие в наборе данных, показаны серым. ( C ) Из 11 дубликатов в Gstu4 – Gste3 , 10 включают Gste2 . На врезке карты показаны страны, в которых не менее 5% людей носили CNV в Gste2 (красный). Черные прямоугольники и вертикальные серые полосы показывают положение генов в кластере, при этом Cyp6aa1 , Cyp6p3 и Gste2 выделены цветом. Фиолетовые горизонтальные полосы показывают степень каждого CNV, а пробел в Gstue_Dup5 показывает удаление в рамках этого расширения. Имена CNV сокращены до Dup # и относятся к Cyp6aap_Dup # и Gstue_Dup # на панелях B и C соответственно.Более подробная информация о каждом из этих CNV, а также о тех из других кластеров генов, представлена в дополнительных данных S5 – S8.
Несколько аллелей CNV были обнаружены в разных популяциях (например, Cyp6aap_Dup7 был обнаружен у An. Coluzzii из Буркина-Фасо, Кот-д’Ивуара, Ганы и Гвинеи) (дополнительные данные S4), хотя ни один из них не был обнаружен во всех популяциях любого вида (дополнительные данные S8).Более того, несколько аллелей CNV, охватывающих одни и те же гены, могут быть обнаружены в одной и той же популяции (например, Cyp9k1_Dup4, Dup11 и Dup15 в An. gambiae из Буркина-Фасо) (Дополнительные данные S7). Для кластера Cyp6aa / p CNV в основном были обнаружены в An. coluzzii из Буркина-Фасо, Кот-д’Ивуара и Ганы (таблица 2). В Cyp9k1 CNV в основном были обнаружены в An. gambiae из Буркина-Фасо, Ганы и Гвинеи.Некоторые гены амплифицировались с очень высокой частотой в определенных популяциях (таблица 2). Например,> 92% от An. gambiae из Буркина-Фасо имел CNV в Cyp9k1 , а> 90% из An. coluzzii из Кот-д’Ивуара содержала гены, покрывающие CNV, в кластере Cyp6aa / p . В целом, 511 из 1142 образцов в этом исследовании (45%) несли хотя бы один из CNV, описанных в этих пяти генах. кластеры.
Таблица 2.Число (и доля) лиц с CNV, охватывающими Cyp6aa1 , Cyp6p3 , Gste2 , Cyp6m2 , Cyp6z1 или Cyp9k1
Мы использовали шаблоны несогласованных чтений, поддерживающих каждый из CNV, чтобы предположительно определить тип события дублирования. что их вызвало (рис.3). Из 44 CNV 33 поддерживаются парным считыванием, отображаемым в геноме, обращенными друг от друга и охватывающими область. увеличенного покрытия, что указывает на тандемное дублирование (Дополнительные данные S4 – S7), и два CNV поддерживались отображением пар чтения в одной ориентации, что указывает на тандемную инверсию (Дополнительные данные S4, S7). Остальные CNV были поддержаны чтениями, чьи спаренные или мягко обрезанные основания отображены в другом месте генома, часто в нескольких геномные местоположения, предполагающие роль мобильных элементов (TE) в событии дупликации (Supplemental Data S4 – S7).Два из этих CNV имели фланкирующие последовательности, которые соответствовали известным мобильным элементам в An. gambiae . Базы чтения с мягким вырезом, отображаемые на точки останова Gstue_Dup7 и Gstue_Dup8, соответственно, вернули значительный BLAST попадает в транспозон ДНК hAT (HATN1_AG; E -value = 2 × 10 −26 ) и транспозон, несущий сходство с элементами Mariner / Tc1-подобными (IKIRARA1; E -value = 10 −34 ). ) (Leung and Romans 1998), в базе данных Repbase (Bao et al.2015).
Рисунок 3.Три типа несогласованных пар чтения ( A – C ) и чтения точки останова ( D ) были использованы для идентификации различных аллелей CNV. ( A ) При тандемном дублировании пары чтения, полученные из сегментов, охватывающих точку останова CNV (красные стрелки), выравниваются лицом от друг друга вокруг точки останова на эталонном геноме (синие стрелки).( B ) В тандемных инверсиях пары считывания, полученные из сегментов, охватывающих конец перевернутого сегмента (красные стрелки), выравниваются по направлению в одном направлении друг с другом вокруг точки останова на эталонном геноме (голубые стрелки). ( C ) При удалении пары чтения, полученные из сегментов, охватывающих удаленную последовательность (красные стрелки), выравниваются в правильной ориентации. вокруг точки останова, но дальше, чем ожидалось, учитывая размер вставки библиотеки секвенирования (зеленые стрелки).( D ) В любом из вышеупомянутых типов CNV (тандемное дублирование показано здесь в качестве примера) считывает пересечение точки останова (красный стрелка) только частично выровняется по обе стороны от точки останова. Для показанного здесь тандемного дублирования начало чтение (светло-коричневая начало стрелки) совпадает с концом дублированной области, тогда как конец считывания (темно-коричневый конец стрелки) выравнивается в начале дублирования.
CNV были дифференцированы на основе их паттернов несогласованных пар чтения, на которые влияет положение Контрольные точки CNV и ориентация дупликации (рис. 3). Возможно, что независимые события дублирования с очень похожими точками останова могут оказаться одним и тем же CNV (например, Cyp6aap_Dup1 может фактически представлять два разных CNV) (Supplemental Data S4), что делает реальное количество событий CNV даже больше, чем сообщается.Точно так же мутация, которая нарушает точку останова CNV может повлиять на его паттерн несогласованных считываний и привести к одному событию CNV, проявляющемуся как два разных аллеля CNV. Хотя мы не можем исключить эту возможность, анализ фона гаплотипов различных CNV показал, что это не случай, по крайней мере, для большинства аллелей CNV (дополнительные данные S8).
CNV в генах метаболической устойчивости проходят положительный отбор
Несколько аллелей CNV были обнаружены с высокими локальными частотами (Supplemental Data S9), что позволяет предположить, что они, вероятно, находятся в процессе положительного отбора.Чтобы исследовать эту возможность, мы поэтапно разделили генотип CNV. обращается к каркасу гаплотипа фазы 2 Ag1000G и вычисляет расширенную гомозиготность гаплотипа (EHH) для аллелей CNV присутствует как минимум у 5% людей в популяции.
Скорость распада EHH вокруг аллелей CNV была постоянно ниже, чем для гаплотипов дикого типа (WT) (рис. 4; дополнительные рис. S6, S8, S10), что подтверждает наше утверждение о том, что эти аллели достигают высокой частоты благодаря положительному отбору.Кроме того, медиана длина общих гаплотипов была значительно выше между парами гаплотипов, несущими один и тот же аллель CNV, чем между гаплотипы дикого типа из той же популяции (бутстрепные 95% доверительные интервалы для медианы не перекрываются) (рис. 5; дополнительные рис. S7, S9, S11).
Рисунок 4.Доказательства длительного неравновесия по сцеплению вокруг CNV в кластере генов Cyp6aa1 – Cyp6p2 .Распад расширенной гомозиготности гаплотипов (EHH) был рассчитан для гаплотипов CNV и не-CNV (WT) с использованием SNP. извне региона, содержащего CNV (разрыв по оси x ): (BF) Буркина-Фасо; (CI) Кот-д’Ивуар; (UG) Уганда; (col) An. coluzzii ; (гам) Ан. gambiae .
Рисунок 5.Длина попарно общих гаплотипов больше между образцами, имеющими аллель CNV, чем между образцами дикого типа. Общий Длины гаплотипов рассчитывали по обе стороны от CNV-содержащей области кластера генов Cyp6aa / p . Образцы, не относящиеся к CNV (WT), были взяты из тех же популяций, что и фокальные аллели CNV. Полоски показывают распределение общих длин гаплотипов между всеми парами гаплотипов с одним и тем же основным гаплотипом.Пределы столбцов показывают межквартильный размах, листовки показывают 5-й и 95-й процентили, горизонтальные черные линии показывают медиану, а выемки на столбцах показывают бутстрапированные 95% доверительный интервал для медианы. Имена CNV (Cyp6aap_Dup #) сокращаются как Dup #: (BF) Буркина-Фасо; (CI) Берег Слоновой Кости; (UG) Уганда; (col) An. coluzzii ; (гам) Ан. gambiae .
Фазирование вызовов генотипа CNV было возможно только для простых дупликаций, когда можно было определить зиготность аллелей CNV от оценки количества копий.Для аллелей CNV с более высоким числом копий (тройное и выше) это было невозможно; таким образом, распад EHH не мог быть рассчитан. В случае Cyp9k1 CNV с самой высокой частотой (Cyp9k1_Dup11, обнаруженный в An. Gambiae из Буркина-Фасо, Ганы и Гвинеи) не может быть фазирован. Поэтому мы исследовали, связана ли эта CNV, на уровень выборки с выбранными гаплотипами. Иерархическая кластеризация гаплотипов в этих трех популяциях выявила два больших кросс-популяционных кластера гаплотипов около Cyp9k1 , что указывает на выборочные развертки (дополнительный рис.S12). Кластер 1 был очень сильно связан с Cyp9k1_Dup11 как у мужчин (точный критерий Фишера, P <0,0001) (дополнительная таблица S3), так и у женщин (ранговая корреляция Спирмена: ρ = 0,9, P <0,0001) (рис. 6A). Кластер 2 был связан с присутствием Cyp9k1_Dup15, но корреляция была не такой сильной, как между кластером 1. и Cyp9k1_Dup11 (ранговая корреляция Спирмена: ρ = 0,65, P <0,0001) (рис. 6B; дополнительная таблица S4).
Рисунок 6.Два основных кластера гаплотипов около Cyp9k1 в Буркина-Фасо, Гане и Гвинее связаны с соответствующими аллелями CNV. Точки дрожат, чтобы показать перекрывающиеся данные. Линии показывают регрессию данных методом наименьших квадратов. ( A ) Сильная корреляция между Cyp9k1_Dup11 и кластером гаплотипов 1.Большинство точек лежат на линии наклона 0,5, что указывает на что Cyp9k1_Dup11 чаще всего встречается как трипликация (две дополнительные копии на хромосому), хотя и более низкие, и более высокие существуют версии этого CNV с номерами копий. ( B ) Более слабая корреляция между Cyp9k1_Dup15 и гаплотипом Cluster 2.
Дупликация
Gste2 в Буркина-Фасо связана с мутацией I114T, вызывающей устойчивость.Десять из 11 аллелей CNV, обнаруженных в кластере Gstu4 – Gste3 , включали Gste2 (рис.2), что, возможно, отражает известную важность этого гена в устойчивости к инсектицидам. Хорошо охарактеризованная мутация I114T в Gste2 , как известно, придает устойчивость к ДДТ (Mitchell et al. 2014) и может быть связан с дупликациями генов аналогично другим мутациям, таким как Ace1 G119S. Поэтому мы исследовали, были ли какие-либо аллели CNV в Gste2 связаны с этой мутацией. Gste2 -114T присутствует по всей Африке, а также в An.gambiae и An. coluzzii ( Anopheles gambiae 1000 Genomes Consortium 2017), но в наших данных был связан только с Gstue_Dup1. Gstue_Dup1 был обнаружен в 16 An. coluzzii образцов из Буркина-Фасо, все из которых были по крайней мере гетерозиготными по 114T (дополнительная таблица S5). Наличие гомозигот 114T вместе с соотношением прочтений, поддерживающих аллели I114 и 114T у гетерозигот (примерно 1: 2), указывают, что обе копии Gste2 в Gstue_Dup1 CNV несут мутацию 114T.
Cyp6aa1 более тесно связан с CNV, чем Cyp6p3Из 15 аллелей CNV, обнаруженных в кластере Cyp6aa1 – Cyp6p2 , пять включали Cyp6p3 , но 13 включали Cyp6aa1 (рис. 2B). Cyp6p3 CNV были обнаружены с высокой (> 50%) частотой в одной популяции (Кот-д’Ивуар An. Coluzzii : 90%), тогда как Cyp6aa1 CNV были обнаружены с высокой частотой в An.coluzzii из Буркина-Фасо (91%), Кот-д’Ивуара (89%) и Гвинеи (75%), а в An. gambiae из Уганды (64%).
Обсуждение
Наше исследование выявило 1557 CNV в 16 популяциях An. gambiae и An. coluzzii , причем CNVs более многочисленны и крупнее в гетерохроматине, чем в эухроматине. Эти параллели приводят к людям и крысам, в котором было обнаружено, что CNV особенно многочисленны в центромерах и теломерах (Nguyen et al.2006; Гурьев и др. 2008), обычно состоящий из гетерохроматина. Распределение CNV по размерам было сильно смещено вправо, при этом небольшие CNV были наиболее распространены как в эухроматине, так и в гетерохроматине, хотя мы не искали CNV размером менее 1500 п.н., чтобы избежать ложных открытия. CNV были смещены в сторону ген-содержащих областей как в целом, так и после исключения гетерохроматиновых областей. Этот смещение для генных областей аналогично было обнаружено при дупликациях у людей (Nguyen et al.2006) и крыс (Гурьев и др., 2008), но не Drosophila (Шрайдер и др., 2016). Таким образом, наши результаты показывают, что изобилие CNV вокруг генов не ограничивается млекопитающими.
Гены, обнаруженные в CNV, сами были обогащены для семей, вовлеченных в метаболическую устойчивость к инсектицидам. Эти результаты зеркальные находки у Drosophila , где цитохром P450 были непропорционально представлены в CNV (Schrider et al.2016). Аналогично в Ae. aegypti , цитохром P450 были обогащены среди генов, демонстрирующих доказательство более высокого числа копий в популяциях, устойчивых к дельтаметрину. по сравнению с восприимчивыми популяциями (Faucon et al. 2015). В нашем исследовании пять метаболических генов, наиболее тесно связанных с устойчивостью к инсектицидам в литературе для An. gambiae и An. coluzzii , которые, как было показано, метаболизируют инсектициды in vitro ( Gste2 , Cyp6p3 , Cyp6m2 , Cyp6z1 и Cyp9k1 ), все они были амплифицированы по крайней мере в одной популяции.Более того, три из этих генов продемонстрировали признаки повторяющегося События CNV внутри и между популяциями, всего 44 различных CNV в пяти кластерах и до 16 в Cyp9k1 . Из 44 CNV большинство (33) были тандемными дупликациями, две – тандемными инверсиями, а две (обе в кластере Gstu4 – Gste3 ) показали связь с известными транспозонами ДНК. Остальные семь не могли быть классифицированы, но наличие неотображаемых последовательностей вокруг точек останова предполагает, что они также могут быть связаны с повторяющимися элементами.
Доказательства важности Gste2 для устойчивости к инсектицидам получены в исследованиях, показывающих его повышение в отношении устойчивости к ДДТ An. gambiae (Динг и др., 2003; Дэвид и др., 2005) и An. funestus (Riveron et al. 2014a) по сравнению с восприимчивыми комарами и трансгенной экспрессией An. gambiae / An. funestus Gste2 в Drosophila , обеспечивающий устойчивость к ДДТ (Mitchell et al.2014; Riveron et al. 2014а). Несинонимичные SNP в Gste2 также были связаны с устойчивостью к ДДТ у An. funestus (Riveron et al. 2014a) и как ДДТ, так и дельтаметрин в An. gambiae (Mitchell et al., 2014; Opondo et al., 2016). В нашем исследовании Gste2 был усилен в Кении, в An. coluzzii из Анголы, Буркина-Фасо и Ганы, а в An. gambiae из Габона и Уганды.
Cyp6p3 активируется у комаров, устойчивых к пиретроидам, ДДТ и бендиокарбу (Djouaka et al.2008; Мюллер и др. 2008; Fossog Tene et al. 2013; Kwiatkowska et al. 2013; Edi et al. 2014; Ngufor et al. 2015), метаболизирует перметрин и дельтаметрин (Müller et al. 2008) и обеспечивает устойчивость к пиретроидам при экспрессии в Drosophila (Edi et al. 2014). Cyp6m2 также активируется у комаров с устойчивостью к перметрину, ДДТ и бендиокарбу (Müller et al. 2007; Djouaka et al. 2008; Mitchell et al. 2012; Edi et al. 2014), метаболизирует пиретроиды и ДДТ ( Стивенсон и др.2011; Mitchell et al. 2012) и обеспечивает устойчивость к пиретроидам, ДДТ и бендиокарбу при экспрессии в Drosophila (Edi et al. 2014). В нашем исследовании было обнаружено, что Cyp6p3 и Cyp6m2 были усилены преимущественно в An. coluzzii из Кот-д’Ивуара, популяция с известной повышающей регуляцией как Cyp6p3 , так и Cyp6m2 по сравнению с восприимчивыми популяциями (Edi et al. 2014). В частности, в случае Cyp6m2 , это повышающее регулирование не может быть вызвано исключительно CNV, потому что частота CNV и количество копий недостаточны для объяснения уровни экспрессии, но давление отбора, направленное на активацию этих генов, могло сыграть роль в поддержании этих CNV в населении.
Cyp6z1 был усилен в An gambiae из Буркина-Фасо и Гвинеи. Cyp6z1 активируется у комаров с устойчивостью к пиретроидам и ДДТ (Nikou et al. 2003; David et al. 2005) и метаболизирует ДДТ и карбарил (Chiu et al. 2008). Наконец, Cyp9k1 был наиболее широко амплифицированным геном из пяти изученных нами кластеров, причем CNV были обнаружены более чем в половине популяций. в нашем наборе данных. Cyp9k1 активируется у комаров, устойчивых к пиретроидам и ДДТ (Fossog Tene et al. 2013; Thomsen et al. 2014; Ngufor et al. 2015), и метаболизирует дельтаметрин (Vontas et al. 2018). Более того, селективное прочесывание в области Cyp9k1 было связано с устойчивостью к инсектицидам у An. coluzzii (Main et al., 2015).
Углубленное исследование CNV вокруг этих пяти генов выявило убедительные доказательства того, что они обеспечивают селективное преимущество.Во-первых, некоторые из аллелей CNV были обнаружены с высокой частотой и в нескольких популяциях. Во-вторых, аллели CNV последовательно продемонстрировали доказательства того, что они находились под положительным отбором, поскольку гомозиготность гаплотипов была расширена для CNV дальше, чем для дикого типа. гаплотипы. Доказательства положительного отбора были также обнаружены в CNV, где оценка EHH не могла быть рассчитана. Cyp9k1_Dup11, который существует как в виде дупликаций, так и в виде троек и, таким образом, не может быть фазирован на каркас гаплотипа для гомозиготности расчет, был последовательно обнаружен в тех же выборках, что и гаплотип большого выборочного свипа около Cyp9k1 в An.gambiae из Буркина-Фасо, Гвинеи и Ганы, что повышает вероятность того, что этот CNV является фокусом выборочной проверки. Хотя Cyp9k1_Dup11 может находиться в неравновесном сцеплении с другими мутациями, высокая частота тройной версии Cyp9k1_Dup11 по сравнению с дублированной версией, обе из которых связаны с развернутым кластером гаплотипов, предполагает, что более высокий порядок Усиления Cyp9k1 обеспечивают избирательное преимущество.Изменения частот аллелей на разных уровнях амплификации в этой CNV потребуют необходимо контролировать, чтобы определить, полностью ли тройное дублирование заменяет дублирование.
В настоящее время одним из основных факторов, рассматриваемых в программах борьбы с малярией, является ценность инвестиций в надкроватные сетки следующего поколения, которые включают пиперонилбутоксид (PBO), которые препятствуют опосредованной цитохромом P450 устойчивости к инсектицидам, и которые были показаны чтобы эффективно снижать численность комаров и заболеваемость малярией по крайней мере в некоторых регионах (Protopopoff et al.2018). Однако в большинстве случаев неясно, является ли местная устойчивость к инсектицидам опосредованной цитохромом P450, частично из-за отсутствие молекулярных маркеров для определения метаболической резистентности. Наши результаты указывают на районы, где возможно появление комаров. для проявления устойчивости на основе цитохрома P450. Например, CNV в кластере Cyp6aa / p в основном были обнаружены в An. coluzzii из Буркина-Фасо, Кот-д’Ивуара и Ганы, тогда как CNV в Cyp9k1 в основном были обнаружены в An.gambiae из Буркина-Фасо, Ганы и Гвинеи. Хотя присутствие CNV цитохрома P450 указывает на вероятное присутствие цитохрома Р450-опосредованная резистентность, мы подчеркиваем, что их отсутствие не обязательно означает, что такой резистентности не существует. Другие формы генетической изменчивости, которые могут увеличивать экспрессию генов, такие как мутации в регуляторной области (Schmidt et al. 2010), нуждаются в исследовании, чтобы получить полный набор молекулярных маркеров для обнаружения метаболической устойчивости.
Все, кроме одного из 11 аллелей CNV в кластере Gstu / e , включали Gste2 , что указывает на то, что это основная цель амплификации гена в этом кластере. Учитывая совокупность доказательств связи Gste2 с ДДТ и устойчивостью к пиретроидам у нескольких видов – An. gambiae (Mitchell et al., 2014), An. funestus (Riveron et al. 2014a), Aedes aegypti (Lumjuan et al. 2011) – фокус амплификации этого гена, вероятно, связан с его важностью для устойчивости.
Дупликация Gstue_Dup1 в Буркина-Фасо происходит на фоне SNP Gste2 _114T, связанного с устойчивостью к ДДТ у An. gambiae (Mitchell et al., 2014). Таким образом, дупликация может служить для увеличения дозировки Gste2 , детоксицирующая активность которого уже повышена мутацией 114T. В качестве альтернативы роль Gstue_Dup1 может быть чтобы компенсировать любые негативные эффекты фитнеса 114T.Хотя нарушение активности Gste2 может быть компенсировано увеличением экспрессии гена, Gstue_Dup1 гомогенен для 114T, исключая возможность компенсации за счет спаривания мутантных и аллелей дикого типа, как обнаружено в гетерогенных дупликациях Ace1 (Assogba et al. 2015).
В кластере Cyp6aa / p только пять из 15 CNV включали Cyp6p3 , и они были обнаружены с заметной частотой только в An.coluzzii из Кот-д’Ивуара. Напротив, 13 из 15 CNV включали Cyp6aa1 , с высокими частотами CNV, обнаруженными в An. coluzzii из Буркина-Фасо, Кот-д’Ивуара и Гвинеи, а в An. gambiae из Уганды. Кроме того, пять высокочастотных CNV, которые включают Cyp6aa1 (Cyp6aap_Dup1, Cyp6aap_Dup7, Cyp6aap_ Dup10, Cyp6aap_Dup14, Cyp6aap_Dup15), все демонстрируют свидетельства положительного отбора. Хотя Cyp6aa1 привлек значительно меньше внимания, чем Cyp6p3 , он ранее был вовлечен в устойчивость к инсектицидам.Экспрессия Cyp6aa1 выше в популяциях An. gambiae и An. coluzzii , которые устойчивы к пиретроидам и ДДТ по сравнению с чувствительными лабораторными колониями (Kwiatkowska et al. 2013; Thomsen et al. 2014). Также имеются убедительные доказательства связи между Cyp6aa1 и устойчивостью к инсектицидам у двух родственных видов. В ан. funestus, экспрессия Cyp6aa1 выше у комаров, переживших воздействие перметрина, по сравнению с чувствительным штаммом (Riveron et al.2014b; Ибрагим и др. 2018), и было показано, что белок метаболизирует пиретроиды и управляет сопротивляемостью при экспрессии в Drosophila (Ibrahim et al. 2018). В ан. minimus , ортолог Cyp6aa1 активируется в результате отбора на устойчивость к дельтаметрину (Rodpradit et al. 2005), и было показано, что белок метаболизирует пиретроиды (Duangkaew et al. 2011). Способность Ан. gambiae Cyp6aa1 для метаболизма инсектицидов эмпирически не тестировалось, хотя теоретическое моделирование предполагает, что это должно эффективно связываются с перметрином и дельтаметрином (Ibrahim et al.2018). Высокая частота амплификаций в Cyp6aa1 и связанные с ними сигналы отбора предполагают, что важность этого гена для устойчивости к инсектицидам у Ан. gambiae и An. coluzzii был недооценен.
В заключение, наши результаты показывают ключевую роль CNVs в адаптивном ответе на сильное и недавнее давление отбора. В популяциях из комаров Anopheles по всей Африке, гены, участвующие в метаболической устойчивости к инсектицидам, были дублированы, и эти дупликации были доведены до высоких частот благодаря положительному отбору.Эти результаты подчеркивают CNV как форму вариации, которая может действовать. как быстрый ответ на селективное давление, требующее изменения уровней экспрессии. Широкое распространение CNV как средства метаболической устойчивости к инсектицидам усиливает необходимость разработки новых инсектицидных соединений для борьбы с перекрестной устойчивостью и подчеркивает потенциальную ценность надкроватных сеток, обработанных ПБО. Наши результаты также выдвигают на первый план Cyp6aa1 как ген, который требует более тщательного изучения на предмет его важности для An.gambiae , которого до сих пор не уделяли должного внимания его геномному соседу Cyp6p3 . В более широком смысле, акцент на SNP в An. gambiae исследования устойчивости к инсектицидам позволили выявить и избирательно распространить мутации числа копий в ключевом инсектициде. гены устойчивости остаются незамеченными. Наши результаты демонстрируют важность наблюдения и расследования CNV в этих гены. С этой целью описания точек останова, представленные в нашем исследовании, позволят проверять и контролировать эти CNV в популяции комаров, что позволяет отслеживать распространение этих мутаций и обеспечивает основу для будущих исследований. исследуя их профиль сопротивления.
Методы
Отбор популяционных проб и полногеномное секвенирование
Мы проанализировали данные по 1142 отдельным выловленным в дикой природе экземплярам An. gambiae и An. coluzzii , собранные и секвенированные на Фазе 2 Ag1000G (https://www.malariagen.net/data/ag1000g-phase-2-ar1) (метаданные представлены в дополнительных данных S10).Экземпляры собраны на стоянках в 13 африканских странах (Ангола An. Coluzzii n = 78, Буркина-Фасо An. Coluzzii n = 75, Буркина-Фасо An. Gambiae n = 92, Камерун An. Gambiae n = 297, Кот-д’Ивуар An. Coluzzii n = 71, Экваториальная Гвинея (Bioko) An. Gambiae n = 9, Габон An. Gambiae n = 69, Гана An. Coluzzii n = 55, Гана An. Gambiae n = 12, Гвинея An. Coluzzii n = 4, Гвинея An.gambiae n = 40, Гвинея-Бисау (смешанное происхождение) n = 91, Кения (неопределенное происхождение) n = 48, Mayotte An. gambiae n = 24, Гамбия (смешанное происхождение) n = 65, Уганда An. gambiae n = 112). Отдельные образцы секвенировали с использованием платформы Illumina HiSeq для получения считываний парных концов 100 пар оснований с мишенью. покрытие 30 ×. Более подробная информация о выборке популяций, подготовке образцов, секвенировании, выравнивании, идентификации видов, и производство данных сообщается в другом месте ( Anopheles gambiae 1000 Genomes Consortium 2017).Метаданные для всех образцов, а также подробные методы сбора для популяций, не включенных в вышеупомянутую публикацию, представлены в дополнительных методах SM1 и дополнительных данных S10.
Расчет и нормализация покрытия
Для каждого человека мы использовали программный пакет pysam (https://github.com/pysam-developers/pysam) для подсчета количества выровненных чтений (покрытия) в неперекрывающихся окнах длиной 300 п.н. по ядерному геному.Положение каждое чтение считалось начальной точкой его выравнивания; таким образом, каждое чтение учитывалось только один раз. Покрытие секвенированием может быть смещены вариациями в локальном нуклеотидном составе. Чтобы учесть это, мы вычислили нормализованное покрытие из чтения подсчет основан на ожидаемом охвате каждого окна с учетом его содержания GC (Абызов и др., 2011). Для каждого окна размером 300 п.н. мы вычислили процент (G + C) нуклеотидов до ближайшей процентной точки в пределах эталона. последовательности, а затем разделили счетчики считываний в каждом окне на среднее количество считываний по всем аутосомным окнам с тем же (G + C) в процентах.Чтобы минимизировать влияние изменения количества копий при вычислении этих нормирующих констант, мы исключили окна из расчета среднего числа считываний, для которых предыдущий анализ доступности генома обнаружил доказательства чрезмерно высокий или низкий охват или неоднозначное выравнивание (окна с доступными основаниями <90% в соответствии с фазой Ag1000G 2 карта доступности генома, именуемая «окнами доступа») (https: // www.malariagen.net/data/ag1000g-phase-2-ar1). Затем нормализованные значения охвата были умножены на коэффициент 2, так что области генома с нормальной диплоидной копией число должно иметь ожидаемое нормализованное покрытие 2.
Прежде чем исследовать нормализованные данные о покрытии для доказательства вариации количества копий, мы применили два фильтра, чтобы исключить окна для которых покрытие может быть ненадежным показателем количества копий.Первый фильтр удалил окна, в которых было выполнено> 2% чтений. выровнено с качеством сопоставления 0 (дополнительный рис. S13), что указывает на то, что считывание отображается неоднозначно и может быть сопоставлено с одинаковым успехом в другом геномном местоположении. Этот фильтр удалил 159,587 (20,8%) из 768,225 окон. Второй фильтр удалял окна, для которых процентное содержание (G + C) был крайним и редко представлен в доступной ссылочной последовательности, то есть менее 100 доступных окон с тот же процент (G + C), потому что небольшое количество окон делает расчет нормирующей константы (G + C) ненадежным.Этот фильтр удалил 13 484 (2,2%) из оставшихся 608 638 окон. Окна, оставленные для анализа, назывались «отфильтрованными» окна. ”
Обнаружение вариаций числа копий в масштабе всего генома
Чтобы определить наиболее вероятное состояние числа копий (CNS) в каждом окне каждого индивидуума, мы применили гауссовский HMM к индивидуальному нормализованные оконные данные о покрытии, аналогичные подходу Miles et al.(2016) и Leffler et al. (2017) (подробнее см. Дополнительные методы SM2). Поскольку нас в первую очередь интересуют амплификации, а не делеции, мы получили необработанный набор вызовов CNV для каждого образец путем обнаружения смежных серий по крайней мере пяти окон с усиленной ЦНС (ЦНС> 2 или ЦНС> 1 для хромосомы X у мужчин).
CNV фильтрация
Из необработанного набора вызовов CNV мы создали отфильтрованный по качеству список вызовов CNV.Сначала мы удалили образцы с очень высоким покрытием. дисперсия, потому что высокая дисперсия может привести к ошибочным вызовам CNV. Таким образом, мы удалили 27 выборок, для которых дисперсия нормализованное покрытие было больше 0,2 (дополнительный рисунок S14), оставив 1115 образцов для дальнейшего анализа.
Затем мы применили два фильтра к необработанным вызовам CNV из этих 1115 выборок. Для первого фильтра мы вычислили вероятность для каждый необработанный вызов CNV как для состояния номера копии, предсказанного HMM, так и для нулевой модели с номером копии = 2, и удален CNV требует, для которых отношение правдоподобия было <1000 (дополнительные методы SM3).Для второго фильтра мы удалили CNV с низкими частотами популяции. Для этого необходимо сопоставить необработанные вызовы CNV. чтобы можно было идентифицировать одну и ту же CNV у разных людей. Мы классифицировали любые два CNV как идентичные, если точки останова предсказанные по их количеству копий, переходы состояний происходили в пределах одного окна друг от друга. Затем мы удалили CNV, которые не были обнаруживается не менее чем у 5% особей как минимум в одной популяции (или как минимум у трех особей для популяций меньше, чем 40).
Мы определили чувствительность и специфичность метода обнаружения CNV, используя моделирование, в котором значения охвата были случайным образом перетасовывались по окнам генома. Подробности этого моделирования описаны в дополнительных данных S11.
CNV в типах хроматина
Области гетерохроматина и эухроматина взяты из Sharakhova et al.(2010). CNV считался гетерохроматическим, если какая-либо его часть перекрывала области гетерохроматина. Мы провели симуляции чтобы определить, были ли обнаруженные нами CNV равномерно распределены между гетерохроматином и эухроматином. За каждый пробег моделирования, мы случайным образом перераспределяли начальные позиции каждого обнаруженного CNV, сохраняя количество отфильтрованных окон покрытые CNV без изменений, и рассчитали количество CNV, которые перекрывают гетерохроматин.Это моделирование было выполнить 10 000 раз, чтобы получить распределение нулевой модели. Двусторонние значения P были получены путем расчета доли моделирования, в результате которого было получено, по крайней мере, столько же гетерохроматических CNV, сколько наблюдаемых в реальных данных и умножая это на два.
Обнаружение дупликаций генов и анализ обогащения генов
Чтобы определить гены, содержащиеся в каждом CNV, мы сравнили начальную и конечную точки CNV с начальной и конечной точками. всех генов, перечисленных в AgamP4.Аннотации 2 генов ( Anopheles-gambiae-PEST_BASEFEATURES_AgamP4.2.gff3 ). Начальные / конечные точки каждого CNV были рассчитаны как медиана начальных / конечных точек всех необработанных вызовов CNV, которые были соответствует этому. Чтобы сохранить только гены, по которым были доступны хорошие данные об охвате, мы оставили только гены, содержащие не менее 50% фильтрованных окон. Мы классифицировали сохраненный ген как скопированный CNV, если все отфильтрованные окна внутри гена находились внутри CNV.Чтобы определить, содержат ли обнаруженные нами CNV больше генов, чем ожидалось случайно, мы провели моделирование. как описано для исследования обогащения для типов хроматина, на этот раз подсчитывая количество CNV, которые содержат не менее один ген. Мы также повторили это моделирование после исключения гетерохроматических областей путем рандомизации только эухроматических областей. CNVs и предотвращение их случайного расположения в гетерохроматине.
Мы определили гены, которые потенциально могут участвовать в метаболической устойчивости посредством детоксикации («гены метаболической детоксикации»). путем нахождения генов, аннотации которых содержали термины «P450», «глутатион-S-трансфераза» или «карбоксилэстераза» в AgamP4 аннотации стенограммы ( Anopheles-gambiae-PEST_ TRANSCRIPTS_AgamP4.2.fa ). Мы провели моделирование, чтобы определить, обогащены ли гены, копируемые CNV, генами метаболической детоксикации.За каждый пробег моделирования, мы рандомизировали каждый генный CNV, перераспределяя гены, охватываемые CNV, сохраняя количество последовательных гены, покрываемые каждым CNV без изменений, и рассчитали количество CNV, которые включали по крайней мере один метаболический детокс-ген. Этот моделирование было выполнено 10 000 раз, чтобы получить распределение нулевой модели. Двусторонние значения P были получены путем расчета доли моделирования, в результате которого было получено, по крайней мере, столько же генных CNV, содержащих детокс гены, наблюдаемые в реальных данных и умноженные на два.
АнализGO генов, включенных в CNVs, был выполнен с использованием пакета topGO в R (https://bioconductor.org/packages/release/bioc/html/topGO.html; R Core Team 2015). Частота ложного обнаружения была рассчитана на основе значений P с использованием пакета R fdrtool (https://cran.r-project.org/web/packages/fdrtool/index.html).
Идентификация аллелей CNV в кандидатных локусах метаболической устойчивости к инсектицидам
Мы подробно охарактеризовали различные события дупликации (аллели CNV) в пяти кластерах генов, представляющих особый интерес ( Cyp6aa1 – Cyp6p2 , Gstu4 – Gste3 , Cyp6m2 – Cyp60003 Cyp6m4 Cyp6z1 , Cyp9k1 ), используя свои уникальные паттерны несогласованных пар чтения и считываний, пересекающих точку останова CNV (считывание точки останова, см.рис.3; Дополнительные методы SM4). Мы вручную проверили пять интересующих областей во всех 1142 выборках, чтобы выявить паттерны несогласованности и точки останова. считывания («диагностические считывания»), последовательно связанные с изменениями в охвате (дополнительные рисунки S2 – S5). Начальная и конечная точки каждого аллеля CNV обычно могут быть точно определены считыванием точки останова, а в противном случае определяется несогласованными парами чтения или точкой изменения покрытия (дополнительные данные S4 – S7).После того, как диагностические считывания были идентифицированы для аллеля CNV, мы зарегистрировали присутствие этого аллеля во всех образцах с не менее двух вспомогательных диагностических чтений.
Чтобы идентифицировать TE, которые могут быть вовлечены в CNV, которые, по-видимому, не являются тандемными инверсиями или тандемными дупликациями, мы взяли мягко обрезанные базы из сопоставления чтения с точками останова этих CNV и использовали несмежные мегабласти для сравнения их против базы данных Repbase (версия 24.10) от Ан. gambiae мобильных и повторяющихся элементов (www.girinst.org/repbase/ [Bao et al. 2015]).
Обнаружение сигналов отбора по аллелям CNV
Мы использовали поэтапные гаплотипы для расчета попарно общей длины гаплотипа и EHH для каждого аллеля CNV (Sabeti et al. 2002), используя только SNP за пределами региона, в котором были обнаружены CNV. Расчеты EHH были выполнены с использованием пакета Python scikit-allel (https: // zenodo.org / record / 3238280).
Кластерыгаплотипов в области Cyp9k1 были получены с scikit-allel с использованием первых 1000 SNP на центромерной стороне Cyp9k1 (теломерная сторона этого гена имеет низкие уровни доступности). Матрица расстояний между гаплотипами была вычислена с использованием доля доступных SNP, которые различались между попарными комбинациями гаплотипов. Это использовалось для выполнения иерархических кластеризация с кластерами гаплотипов, определенными с использованием порогового значения 0.001.
Статистика
Статистический анализ проводился в R (R Core Team 2015). Таблицы непредвиденных обстоятельств были проанализированы с помощью точного критерия Фишера. Если размер выборки был слишком большим для точного теста Фишера, P -значения были получены с использованием опции «simulated.p.value» с 10 6 повторами.
Оценка числа копий, специфичных для аллелей, и фазирование генотипов CNV на каркасы гаплотипов Ag1000G
Чтобы определить количество аллель-специфичных копий в образце, мы оценили изменение покрытия, связанное с каждым аллелем CNV. (Дополнительный рис.S15; Дополнительные методы SM5). Таким образом, даже когда перекрывающиеся аллели CNV присутствовали в одном образце, мы обычно могли определить количество копии каждого аллеля.
Для однокопийных CNV можно определить генотип образца по количеству копий (число копий 1 указывает гетерозигота, число копий 2 указывает на гомозиготу по CNV). Для CNV более высокого порядка это невозможно, потому что дупликацию гетерозиготы нельзя отличить от дупликации гомозиготы.Поэтому мы применили два фильтра, чтобы сохранить только однокопийные аллели CNV. Первый фильтр удалил аллели CNV, для которых было обнаружено, что число копий, специфичных для аллелей, подняться выше 2 в данных (если только один образец поднялся до 2,5, мы предположили, что это может быть ошибкой, и классифицировали это как 2). Этот фильтр удалил пять аллелей CNV (Cyp6aap_Dup11, Gstue_Dup2, Gstue_Dup8, Cyp9k1_Dup11, Cyp9k1_Dup15). Для второй фильтр, мы классифицировали каждый образец как гомозиготу дикого типа, гетерозиготу или гомозиготу CNV на основе их количества копий, а затем удалили аллели CNV, которые не соответствовали ожиданиям Харди-Вайнберга в популяциях, в которых они были найдены.Этот фильтр удалил четыре аллеля CNV (Cyp6aap_Dup4, Gstue_Dup5, Gstue_Dup7, Cyp9k1_Dup10). Три аллеля CNV (Cyp9k1_Dup7, Cyp9k1_Dup13 и Cyp9k1_Dup14) также были исключены из-за трудностей с определением аллель-специфичного номера копии (Supplemental Data S7). В одном случае (Cyp6m_Dup1) было обнаружено, что у всех людей число копий равно 2, что указывает на то, что CNV является троекратным, без дублирования в популяции. Таким образом, этот CNV был сохранен, и все образцы, содержащие CNV, были классифицированы. как гетерозиготный.
АллелиCNV, прошедшие оба фильтра, были фазированы на каркасы гаплотипов Ag1000G Phase 2 с использованием программного обеспечения MVNcall v1.0. (Menelaou and Marchini 2013), используя параметры по умолчанию, кроме установки λ = 0,1, чтобы гарантировать, что ни один из входных вызовов генотипа CNV не был изменен во время фазирования. Для каждого из пяти кластеров генов фазирование было выполнено с использованием 200 неядерных SNP по обе стороны от области, в которой были обнаружены CNV, что позволило избежать включение SNP, найденных в любой из CNV.Гаплотипы, содержащие более одного аллеля CNV, были редкими и, следовательно, исключены из последующих расчетов гомозиготности гаплотипов.
Благодарности
Мы благодарим трех анонимных рецензентов за полезные комментарии к этой рукописи. Эта работа была поддержана Wellcome Trust (0 / Z / 09 / Z; 0 / Z / 09 / Z; 098051), Совет медицинских исследований Соединенного Королевства (MR / P02520X / 1; MR / M006212 / 1) и Национальный институт Аллергия и инфекционные заболевания ([NIAID] R01-AI116811).Авторы полностью несут ответственность за содержание и не обязательно представляют официальную точку зрения NIAID или Национальных институтов здравоохранения (NIH).
Вклад авторов: E.R.L., A.M., M.K.N.L., D.P.K., D.W. и M.J.D. разработал исследование. E.R.L., A.M., N.J.H. и C.S.C. провели анализ. Консорциум Ag1000G провел сбор, подготовку, секвенирование и первичный анализ образцов.E.R.L., A.M., D.W., и M.J.D. написал рукопись. Все авторы прочитали и одобрили окончательную рукопись.
- Поступило 30.10.2018 г.
- Принята к печати 26 июня 2019 г.
Какие варианты количества копий?
У каждого человека есть уникальные варианты в своем геноме, и многие из них безвредны, но какие типы могут влиять на наше здоровье и как они возникают?
Большие части генома человека состоят из повторяющихся последовательностей.Некоторые из них представляют собой короткие последовательности из двух или трех пар оснований («буквы» ДНК), повторяющиеся десятки или сотни раз; другие могут включать дупликации целых генов или более крупных участков хромосом. Варианты числа копий (CNV) – это случаи, когда количество повторов варьируется от человека к человеку и может составлять почти 10% генома человека.
Многие из этих вариантов, по-видимому, не влияют на здоровье, но некоторые связаны с болезнью или могут иметь другие клинически значимые эффекты. Два типа CNV – тринуклеотидные повторы и дупликации целых генов – могут иметь особенно большое влияние на здоровье пораженных людей.
Тринуклеотидные повторы и болезнь
Одним из наиболее известных примеров вызывающей заболевание CNV является болезнь Хантингтона, которая вызывается повторяющейся последовательностью из трех пар оснований (известной как тринуклеотидный повтор) в конце кодирующей области гена HTT . .
У здоровых людей последовательность «CAG» повторяется от 10 до 35 раз, но люди с 40 или более повторами будут поражены болезнью Хантингтона. Возможно большое количество повторов (более 200), а большее количество повторов, особенно более 60, связано с более ранним началом заболевания.Болезнь показывает пониженную пенетрантность у людей с 36 до 39 повторами.
Тринуклеотидные повторы также ответственны за синдром ломкой Х-хромосомы и связанные с ним состояния (известные как «предварительные мутации»), которые возникают из-за повтора «CGG» в некодирующей области гена FMR1 .
У здоровых людей будет от 5 до 40 повторов, но состояние здоровья проявляется у людей с большим количеством повторов. Те, у кого от 55 до 200 повторов, имеют преждевременную мутацию и подвержены риску различных клинических расстройств, включая проблемы развития нервной системы, психические расстройства и преждевременную менопаузу.Те, у кого более 200 повторов, имеют синдром ломкой Х-хромосомы. Такое количество повторов приводит к инактивации промотора гена и выключению гена, даже если кодирующие области остаются неизменными.
Дупликации целых генов: значение для здравоохранения
CNV также могут включать в себя повторение целых генов в геноме. Одним из примеров является ген CYP2D6 , который кодирует цитохром P450 у человека. P450 – это фермент, важный для расщепления веществ, не вырабатываемых организмом, в том числе лекарств.
Наряду с наличием нескольких различных аллелей, которые могут вызывать вариации активности P450, у некоторых людей также есть дупликации этого гена, что может сделать их сверхвысокими метаболизаторами. Это может иметь значительные клинические последствия, поскольку эти люди будут реагировать на дозы лекарств совершенно иначе, чем другие в популяции.
Во многих случаях сверхвысокие метаболизаторы расщепляют лекарства быстрее, поэтому обычная дозировка будет неэффективной для пациента.P450 участвует в метаболизме нескольких антидепрессантов и нейролептиков, что делает эту область интересной при назначении психиатрических препаратов.
Однако некоторые препараты, такие как кодеин, используют P450. Это соединения (известные как пролекарства), которые организм превращает в активную форму – в случае кодеина – в морфин. Для сверхвысоких метаболизаторов этот эффект усиливается, так что в конечном итоге у пациента может быть гораздо более высокая концентрация морфина в кровотоке, чем предполагал врач.
Чтобы узнать больше о типах геномных вариантов, которые могут повлиять на здоровье человека, прочитайте нашу поясняющую статью «Различные типы вариантов: что такое геномные вариации?»
–
Обратите внимание: эта статья предназначена для информационных или образовательных целей и не заменяет профессиональную медицинскую консультацию.Вариация числа копий гена не отражает структуру или экологический отбор в двух недавно разошедшихся калифорнийских популяциях Suillus brevipes | G3: Гены | Геномы
Abstract
Было показано, что вариация числа копий гена у разных людей отслеживает структуру популяции и является источником адаптивных генетических вариаций со значительным влиянием на приспособленность.В этом исследовании мы сообщаем о противоположных результатах для обоих прогнозов, основанных на анализе вариантов числа копий гена (CNV) Suillus brevipes , микоризного гриба, адаптированного к прибрежным и горным местам обитания в Калифорнии. Чтобы оценить, отражают ли вариации числа копий гена структуру популяции и отбор у этого вида, мы исследовали две ранее изученные локально адаптированные популяции, показывающие высокодифференцированную геномную область, включающую ген, который, по прогнозам, придает солеустойчивость.Кроме того, мы исследовали, различалось ли количество копий в генах, связанных с солевым гомеостазом, между двумя популяциями. Хотя мы обнаружили множество примеров областей CNV в геномах особей S. brevipes , мы также обнаружили, что CNV не восстанавливают структуру популяции, и известные гены, связанные с солеустойчивостью, не подвергались отбору среди прибрежной популяции. Наши результаты контрастируют с предсказаниями CNV, совпадающими с дивергенцией однонуклеотидного полиморфизма, и показали, что CNV генов солевого гомеостаза не подвергаются отбору в S.Крестьянка .
Варианты числа копий (CNV) – это гены или области генома различного размера с различным числом копий у разных людей. Различия в количестве копий могут возникать из-за геномных перестроек (таких как неаллельная гомологичная рекомбинация и негомологичное соединение концов (Gu et al. 2008; Conrad et al. 2010; Lupski and Stankiewicz 2005), остановка вилок репликации (Hull et al. 2017) и циркуляризация ДНК (Møller et al. 2015).CNV могут быть специфичными для популяции и иметь большое влияние на динамику популяции. Например, было показано, что отбор эффективно удаляет вредные CNV в больших популяциях малярийных паразитов и менее эффективно – в меньших популяциях (Cheeseman et al. 2015). Структура популяции и истории отбора могут быть восстановлены с помощью анализа CNV на уровне популяции. Было показано, что у людей CNV отражают структуру популяции, обнаруженную по дивергенции однонуклеотидных полиморфизмов (SNP) (Jakobsson et al. 2008; Конрад и др. 2010; Редон и др. 2006). Несмотря на то, что CNV отражают паттерны популяционной структуры, полученные из однонуклеотидных полиморфизмов (SNP) у многих организмов, включая человека, в траве Brachipodium distachyon (Gordon et al. 2017), у грибковых патогенов Zymoseptoria tritici и Asum. (Hartmann, Croll, 2017; Zhao, Gibbons, 2018), у коренного китайского крупного рогатого скота, адаптированного к высокогорью (Zhang et al. 2020), это не всегда так. Например, исследование популяции американских омаров через градиент температуры на входе, показывающее отсутствие структуры популяции на основе SNP, показало, что CNV являются лучшими предикторами отбора, чем SNP, и несоответствие между паттерном, полученным с помощью SNP и CNV (Dorant et al. 2020).
Обнаружение CNV у индивидуумов важно, потому что варианты, вызывающие резкие изменения фенотипов, могут быть адаптивными и иметь важные эволюционные последствия.Отбор может модулировать дозировку белка (где увеличенное число копий приводит к увеличению количества продукта гена в клетке) или вызывать функциональную дивергенцию между копиями генов, приводящую к дифференцированно адаптивным фенотипам (Schrider and Hahn 2010; Kondrashov 2012). CNVs и, в частности, размножение генов, как известно, важны для адаптации к окружающей среде (Oh et al. 2012). Известно, что вариация числа копий генов участвует в адаптации к окружающей среде, характеризующейся экстремальными температурами, токсичными тяжелыми металлами и высокой соленостью (Кондрашов, 2012; Oh et al. 2012). Например, расширение семейства генов гликопротеинов-антифризов позволяет треске выдерживать отрицательные температуры в Антарктике (Chen et al. 2008), а увеличенное количество копий генов, связанных с термостойкостью, придает термостойкость штаммам Escherichia coli ( Риле и др. 2001). Эволюция толерантности к тяжелым металлам также может происходить через CNV (Oh et al. 2012), включая увеличение количества копий и уровней экспрессии генов гомеостаза металлов у растений (Craciun et al. 2012; Talke et al. 2006) и грибов (Ruytinx et al. 2019; Bazzicalupo et al. 2020). Толерантность к соли также может быть связана с CNV гена-переносчика ионов и была зарегистрирована у нескольких растений (Huang et al. 2008; Wu et al. 2012) и у дрожжей Saccharomyces cerevisiae , где повышенные уровни плоидности и экспрессия связаны с толерантностью к высокой солености (Dhar et al. 2011).
У грибов отсутствуют легко поддающиеся измерению фенотипы, что затрудняет изучение адаптации грибов к окружающей среде (Branco et al. 2017; Бранко 2019). Одним из распространенных подходов к обнаружению геномных признаков отбора является использование как сканирования генома для отбора, так и корреляций между геномами и окружающей средой, что подчеркивает важность конкретных генов в способности грибов сохраняться в определенных средах. По большей части этот подход чаще всего применялся к данным SNP (Ellison et al. 2011; Gladieux et al. 2015; Branco et al. 2015; Branco et al. 2017). Однако, учитывая эволюционную важность CNV, сканирование генома, нацеленное на вариацию числа копий, также было полезно для обнаружения адаптивных вариантов у грибов, таких как винные штаммы дрожжей Saccharomyces cerevisiae , патоген человека Cryptococcus gattii и микоризный гриб . Suillus luteus (Steenwyk, Rokas, 2017; Steenwyk, Rokas, 2018; Steenwyk, et al. 2016; Bazzicalupo et al. 2020).
Здесь мы исследуем, отражает ли вариация числа копий гена паттерны структуры популяции и отбора, выявленные при сканировании генома по расхождению SNP у Suillus brevipes , широко распространенного микоризного гриба, связанного с соснами. В предыдущей работе были задокументированы дифференцированные популяции из прибрежных и горных регионов Калифорнии, адаптированные к различным климатическим условиям и условиям солености (Branco et al. 2015; Branco et al. 2017), с дифференцировкой SNP, выявляющей сильную сигнатуру отбора около гена, связанного с солеустойчивостью (предсказанный как Nha-1 -подобный, Na + / H + -антипортер). Кроме того, не было обнаружено вариаций SNP в этом гене у прибрежных особей, что убедительно свидетельствует о селективном поиске адаптивных аллелей, позволяющих колонизировать соляную среду в прибрежной Калифорнии. Сообщается, что гомологов Nha-1 важны для толерантности к соли у других организмов, включая повышенные уровни экспрессии Nha-1 у Arabidopsis thaliana , что позволяет выжить при высоких концентрациях натрия (Apse et al. 1999) и Nha-1 мутаций, передающих солеустойчивость у дрожжей (Nass et al. 1997). Кроме того, имеются данные о солеустойчивых штаммах дрожжей, имеющих множественные копии Nha-1 (Prior et al. 1996). Мы предположили, что CNV в двух популяциях S. brevipes отражают как популяционную структуру, так и модели отбора, полученные из анализа SNP, и преобладают в определенных геномных регионах, связанных с солевым гомеостазом. В частности, мы исследовали, восстанавливают ли CNV ранее описанную дифференциацию популяций между прибрежной и горной Калифорнией и участвуют ли наиболее дифференцированные CNV гены солевого гомеостаза, включая Nha-1 -подобный ген.Мы обнаружили, что у грибов обнаруживается много CNV, однако эти CNV не смогли воспроизвести популяционную структуру S. brevipes и признаки отбора, предсказанные анализом SNP.
Методы
Оценки числа копий гена на индивидуальную и популяционную дифференциацию числа копий гена
Мы оценили вариацию числа копий гена на основе обработанных считываний Illumina 27 полных геномов S. brevipes из прибрежных (11 особей) и горных ( 16 человек) Популяции Калифорнии, ранее проанализированные в Branco et al. (2015). Считанные данные доступны в GenBank (см. Бранко и др. (2015) для кодов SRA отдельных лиц и эталонного генома). Чтобы обнаружить вариацию числа копий в популяциях, мы оценили количество копий для окон 250 пар оснований у каждого человека с помощью программного обеспечения Control-FreeC (Barillot et al. 2011). Suillus brevipes – дикариотический гриб, что означает, что в одной и той же клетке обитают два гаплоидных генома, поэтому мы предположили диплоидность наших образцов и последовали примеру Steenwyk et al. (2016) для других настроек программного обеспечения. Control-FreeC сопоставляет считанные данные с эталонным геномом, нормализует их, предполагая диплоидность, и оценивает отклонения на основе количества считываний. Только гены, присутствующие в эталоне, оцениваются на предмет отклонений числа копий, а гены, отсутствующие в образце, считаются потерями. Мы определяем «выигрыш» как регионы, которые, по оценкам, имеют более двух копий, и «потери» или «отсутствия» как регионы, которые, по оценкам, имеют одну или ноль копий гена. Этот метод, основанный на нормализованном количестве считываний, не позволяет нам по-настоящему оценить, находятся ли копии определенного гена в тех же участках генома или в других частях генома.Чтобы идентифицировать регионы, дифференцированные по количеству копий, мы вычислили величину дисперсии копий, используя V ST после Steenwyk et al. (2016) (код с формулой V ST доступен по адресу https://github.com/abazzical/sbrevCNV). Учитывая, что эталонный геном довольно фрагментирован (Branco et al. 2015), мы приводим результаты только для 100 крупнейших каркасов генома для всех последующих анализов. Общая длина референсного генома составляет 52 222 250 п.н., а скаффолды 1-100 представляют 54.7% (28 583 750 б.п.). Поскольку каркасы в эталонных геномах пронумерованы в соответствии с их размером, мы наблюдали увеличение значений V ST по мере уменьшения размера каркаса, что, возможно, указывает на некоторую систематическую ошибку в оценке числа копий, когда эталонный каркас более фрагментирован. Поэтому мы решили использовать только первые 100 каркасов (рис. S1-2).
Кластеризация индивидуумов на основе полногеномных вариантов числа копий (CNV)
Чтобы выяснить, восстанавливает ли вариация числа копий дифференциацию популяции, обнаруженную в (Branco et al. 2017; Бранко и др. 2015), мы провели анализ главных координат (PCoA) на основе оценок числа копий гена. Цель состояла в том, чтобы оценить, сгруппированы ли отдельно представители прибрежных и горных популяций, как видно из данных SNP (Branco et al. 2015; Branco et al. 2017). С этой целью мы использовали матрицы оценок количества копий гена для каждого отдельного гена в окнах 250 п.н. по всем образцам с помощью программного обеспечения Bedtools (Quinlan and Hall 2010; Quinlan 2014).Мы составили четыре матрицы расстояний, отфильтровав исходную матрицу разными способами. Одна матрица включала все CNV областей кодирующей последовательности ДНК (CDS), вторая включала только верхний 1% дифференцированных CDS. Мы также отфильтровали матрицы на основе количества копий: третья матрица включала только области, которые имели хотя бы одно «усиление», а четвертая матрица включала только области, которые имели хотя бы одно нулевое значение. Все матрицы расстояний и PCoA были сгенерированы с использованием пакета ape (Paradis and Schliep 2018) в R (R Core Team 2014).Для сравнения размеров и количества горных и прибрежных CNV мы использовали критерий суммы рангов Вилкоксона. Кроме того, чтобы визуализировать, насколько люди похожи друг на друга, мы использовали матрицу расстояний, используемую в PCoA для всех генов с CNV, чтобы построить кластерное дерево с настройками по умолчанию R package ape (Paradis and Schliep 2018). . Затем мы смогли сравнить наше дерево кластеризации с деревом штата Нью-Джерси на основе SNP из (Branco et al. 2015).
Обогащенный анализ онтологии генов для 1% наиболее различающихся CNV
Чтобы оценить, были ли гены, дифференцированные по верхнему CNV, обогащены генами, участвующими в солеустойчивости, мы провели анализ обогащения онтологии генов (GO) на 1% верхних отклоненных CNV между два S.brevipes популяций. Мы использовали термины GO на основе аннотированного эталонного генома S. brevipes (Бранко и др. 2015) и использовали приложение ClueGO (Кириловский и др. 2009) в Cytoscape (Шеннон и др. 2003) для определения важных терминов GO и описания функций генов. Мы установили значимость обогащения терминов GO путем реализации двустороннего гипергеометрического теста и скорректированных Бенджамини – Хохберга p-значений с использованием статистики ClueGO.
Представляющие интерес гены, связанные с солевым гомеостазом
Для идентификации генов, связанных с солеустойчивостью, мы использовали эталонный геном Suillus brevipes (Branco et al. 2015) из базы данных MycoCosm (Григорьев и др. 2013). Мы провели поиск в базе данных по ключевым словам «Na + » и «натрий». Количество копий генов было восстановлено с помощью Bedtools (Quinlan 2014), и мы построили тепловые карты, отображающие количество копий генов в R (R Core Team 2014), используя gplots (Warnes et al. 2009) и Rcolorbrewer (Neuwirth 2014).
Мы выполнили точные тесты Фишера (R Core Team 2014), чтобы выяснить, существует ли значительная разница в количестве копий гена, связанного с солевым гомеостазом, между популяциями.Для каждого человека количество копий выбранных генов оценивали как усиление, отсутствие или нейтральность на основе эталонного генома. Точные тесты Фишера были выполнены с ожидаемым отношением шансов 1: 1 и доверительным интервалом 95%. Мы использовали поправку Бонферрони для множественного тестирования. Весь код доступен на https://github.com/abazzical/sbrevCNV.
Результаты
CNV генов не отражают структуру SNP у Suillus brevipes
В отличие от предыдущих результатов SNP, показывающих четкую дифференциацию популяции (Branco et al .2015), не было значительных различий в количестве CNV или размере CNV между горными и прибрежными популяциями (критерий суммы рангов Вилкоксона P > 0,05). Подавляющее большинство CNV были потеряны по всему геному (Таблица 1). Около 10% CNV были локализованы в предполагаемых генах всех изолятов. В соответствии с результатами, полученными на других грибах (Hartmann and Croll, 2017), мы обнаружили гораздо меньшее увеличение числа копий по сравнению с потерями как по геному, так и по прогнозируемым генам. Потери были в среднем гораздо большими участками генома по сравнению с размером дупликаций / умножений (Таблица 1, Рисунок 1).
Таблица 1 Число вариантов числа копий и средний размер для всех индивидуумов для всего генома и только регионов, прогнозируемых как гены толерантность. A и B – это размеры по всему геному, C и D – размеры для предсказанных генов. A и C – это горная популяция, а B и D – прибрежная популяция.Мы также обнаружили, что CNV не демонстрируют ту же картину, что и SNP, и не смогли восстановить структуру популяции прибрежных и горных особей S.brevipes в Калифорнии. Этот результат был очевиден как из анализа всего генома CNV PCoA (Рисунок 2), так и из кластерного анализа (Рисунок S4). Ни один из анализов не показал различий между двумя популяциями, поскольку люди не группировались на основе их происхождения. Мы обнаружили вариации в количестве копий генов у разных людей, как распределенных по геному, так и генов, связанных с солеустойчивостью (см. Раздел ниже). При рассмотрении только областей, показывающих «прирост» или «отсутствие», мы обнаружили, что ординации неразличимы, что предполагает, что существует очень небольшая общая разница в потерях и дупликациях генов (рис. S5 и S6).
Рисунок 2Варианты количества копий гена не отслеживают структуру популяции Suillus brevipes . Анализ основных координат вариантов числа копий полногенома для S. brevipes особей из прибрежных (черные кружки) и горных (пустые кружки) популяций в Калифорнии.
Верхние дифференцированные области CNV не обогащены генами, связанными с солевым гомеостазом
Вариация числа копий гена была распределена по геному, при этом некоторые области демонстрируют гораздо большую дифференциацию по сравнению с другими (Рисунок 3).PCoA, основанный на 1% наиболее дифференцированных генов CNV, показал, что прибрежная популяция сгруппирована в плотную группу, что можно объяснить более сильным отбором или недавним узким местом прибрежной популяции, описанным в (Branco et al. 2015) (рис. S1 ). Верхние 1% окна размером 250 п.н. перекрывались с 37 537 аннотациями из эталонного генома JGI и включали 16 472 «CDS», 18 362 «экзон», 1347 «стоп-кодон» и 1355 «стартовый кодон». Вопреки нашим ожиданиям, верхний 1% CNV не был обогащен генами, которые, как известно, связаны с солеустойчивостью.Обогащенный анализ генной онтологии для 1% дивергированных генов выявил «убиквитин» как единственный значительно представленный термин GO (Таблица S1). Убиквитин присутствует у эукариот и регулирует белки в клетке, и неясно, как функция этого гена связана с адаптацией к окружающей среде или соли в двух популяциях S. brevipes (Hershko and Ciechanover 1998).
Рисунок 3Suillus brevipes прибрежная / горная геномная дивергенция на основе значений V ST (разница в дисперсии числа копий между популяциями (Redon et al. 2006). Анализ проводился на окнах размером 250 п.н. самых больших референсных матриц генома 100. Точки синего цвета представляют верхний 1% значений V ST и обогащены убиквитином.
Отсутствие дифференциации числа копий в генах, специфичных для солеустойчивости, между прибрежными и горными популяциями Suillus brevipes
Мы обнаружили 83 гена, участвующих в солевом гомеостазе в геноме S. brevipes , включая «белки-обменники натрия / кальция», Na + / дикарбоксилат, Na + / переносчики трикарбоксилата и фосфата и «Семейство натрий / водородообменников».Основываясь на точных тестах Фишера, ни один из этих генов не показал значительных различий в CNV между прибрежными и горными популяциями (Таблица S2), предполагая, что гены солевого гомеостаза не подвергаются отбору по разному количеству копий в этих двух популяциях. Однако, как показано на тепловой карте на рисунке 4, количество копий варьируется у разных людей в обеих популяциях.
Рисунок 4Вариация числа копий гена в генах Suillus brevipes , участвующих в солевом гомеостазе. На тепловой карте показано изменение количества копий гена для 83 задействованных генов.Цветовая шкала от бордового до синего, переходящая в желтые, соответствует числам от «0» (бордовый) до «12» (синий). (Особи 1-18 относятся к горной популяции, а 20-34 – к прибрежной). Ген Nha- обозначен стрелкой «<». Идентификаторы белков каждого гена указаны в правой части графика.
Ген, связанный с солью, идентифицированный Branco et al. (2015) ( Nha-1 -подобный ген; ProteinID 1095265) показал диапазон числа копий у людей (рисунок 4).Однако не было согласованного паттерна между двумя популяциями, что указывает на отсутствие отбора по количеству копий Nha-1 -подобного гена.
Обсуждение
Мы обнаружили, что CNV не восстанавливают паттерны популяционной структуры и отбора, ранее задокументированные в Suillus brevipes . Известно, что этот вид генетически дифференцирован в прибрежной и горной Калифорнии и демонстрирует геномные признаки местной адаптации к высокой засоленности почвы (Branco et al. 2015). Вопреки нашим ожиданиям, мы не обнаружили доказательств дифференциации вариаций числа копий как по геному, так и по генам, связанным с солеустойчивостью. Однако индивидуумы в обеих популяциях показали CNVs в своих геномах, что согласуется с вариацией числа копий в рибосомном внутреннем транскрибируемом спейсере для особей одного и того же вида лишайниковых грибов (Bradshaw et al. 2020).
Учитывая предыдущие результаты в S. brevipes , мы предположили, что CNV, участвующие в солевом гомеостазе, находятся под отбором, особенно Nha-1 -подобный ген, который, как было ранее задокументировано, находится под положительным отбором.Люди в пределах популяции имеют общую демографическую историю, которая влияет как на SNP, так и на вариацию числа копий гена, поэтому разумно ожидать, что оба типа генетической вариации будут демонстрировать сходные модели структуры популяции и отбора. Фактически, такие примеры хорошо задокументированы в человеческих популяциях как по всему геному (Jakobsson et al. 2008), так и по конкретным генам, связанным с функцией врожденного иммунитета (Ballana et al. 2007). Имеются также случаи совпадения паттернов SNP и CNV у грибов, в том числе у патогена растений Zymoseptoria tritici , где CNV генов и дивергенция SNP демонстрируют одинаковую структуру популяции во всем мире (Hartmann and Croll 2017; Zhao and Gibbons 2018).
Как биологические, так и технические факторы, вероятно, способствовали отсутствию соответствия между CNV и SNP у S. brevipes , включая недавнее расхождение популяций, слабый отбор и сильно фрагментированный эталонный геном. Несмотря на то, что прибрежные и горные популяции S. brevipes четко различаются и изолированы из-за прерывистого присутствия хозяев сосны в Центральной Калифорнийской долине, они обладают значительным количеством наследственных генетических вариаций, указывающих на довольно недавнее разделение (Branco et al., 2015), что, возможно, еще не позволило установить дифференциацию CNV. Кроме того, слабый естественный отбор также потенциально может способствовать отсутствию сигнала CNV. Несмотря на то, что есть четкие доказательства местной адаптации у S. brevipes из Калифорнии (Branco et al. 2015, 2017), ранее обнаруженная геномная сигнатура солеустойчивости была ограничена одной областью, включая один ген, который, как известно, участвует в солевой гомеостаз, предполагающий, что засоление почвы не может быть очень сильным селективным давлением для этого вида.Прибрежные и горные популяции S. brevipes были отобраны из мест с отличным химическим составом почвы (Peay et al. 2010), и отсутствие генетического разнообразия в гене Nha-1 в прибрежной популяции было хорошим показателем. засоления почвы является селективным давлением для S. brevipes (Бранко и др. 2015). Однако возможно, что содержание солей в прибрежных калифорнийских почвах недостаточно высоко, чтобы приводить к генетическим признакам адаптации CNVs.И наоборот, отбор по связанным с солью CNVs также может быть слабым, потому что прибрежный вариант гена транспорта натрия, обнаруженный Branco et al. (2015) достаточно для обеспечения преимущества в прибрежной среде. Наконец, S. brevipes не является модельным организмом и имеет ограниченные доступные геномные ресурсы, которые могли бы помешать восстановлению более сильного сигнала CNV. Эталонный геном для этого вида сильно фрагментирован (собран в 1550 каркасов), а его аннотация неполна и основана на предсказаниях генов на основе сходства последовательностей (Kuo et al. 2014). Эти ресурсы позволяют раскрыть важные аспекты биологии S. brevipes , но могут помешать обнаружению ключевых генов, участвующих в адаптации к окружающей среде. Мы исследовали ~ 52% генома (включенного в 100 крупнейших каркасов), и могли быть адаптивные CNV, связанные с солеустойчивостью в остальной части генома, которые остались необнаруженными.
В заключение, наши результаты дополняют результаты предыдущих исследований адаптации к окружающей среде микоризного гриба S.brevipes (Бранко и др. 2017; Бранко и др. 2015) и в более широком плане вносят свой вклад в ранее незарегистрированный сценарий CNV, не отражающий паттерны структуры населения или отбора, отображаемые вариацией SNP.
Благодарности
Мы хотели бы поблагодарить Jennifer Lachowiec за полезные комментарии к рукописи.
- Получено 10 июля 2020 г.
- Принято 12 октября 2020 г.
- Авторские права © 2020 Bazzicalupo et al.
Это статья в открытом доступе, распространяемая в соответствии с условиями Международной лицензии Creative Commons Attribution 4.0 (http://creativecommons.org/licenses/by/4.0/), которая разрешает неограниченное использование, распространение и воспроизведение в на любом носителе при условии правильного цитирования оригинальной работы.
% PDF-1.5 % 799 0 объект > эндобдж xref 799 130 0000000016 00000 н. 0000005071 00000 н. 0000005238 00000 п. 0000005830 00000 н. 0000006318 00000 н. 0000007039 00000 п. 0000007527 00000 н. 0000007641 00000 н. 0000007738 00000 н. 0000008357 00000 н. 0000009034 00000 н. 0000009127 00000 н. 0000009704 00000 н. 0000010564 00000 п. 0000011350 00000 п. 0000011763 00000 п. 0000011913 00000 п. 0000011976 00000 п. 0000012413 00000 п. 0000013272 00000 п. 0000013714 00000 п. 0000013799 00000 п. 0000014173 00000 п. 0000014666 00000 п. 0000015585 00000 п. 0000015751 00000 п. 0000016063 00000 п. 0000016392 00000 п. 0000017288 00000 п. 0000017454 00000 п. 0000017569 00000 п. 0000018721 00000 п. 0000019069 00000 п. 0000020056 00000 п. 0000020932 00000 п. 0000023034 00000 п. 0000023823 00000 п. 0000023906 00000 п. 0000023961 00000 п. 0000029102 00000 п. 0000032267 00000 п. 0000034093 00000 п. 0000037019 00000 п. 0000040871 00000 п. 0000040995 00000 п. 0000041111 00000 п. 0000041189 00000 п. 0000041382 00000 п. 0000041460 00000 п. 0000041653 00000 п. 0000041731 00000 п. 0000044232 00000 п. 0000044310 00000 п. 0000046859 00000 н. 0000046937 00000 п. 0000049239 00000 п. 0000049317 00000 п. 0000051978 00000 п. 0000052056 00000 п. 0000054688 00000 п. 0000054766 00000 п. 0000057482 00000 п. 0000057560 00000 п. 0000060204 00000 п. 0000060282 00000 п. 0000062813 00000 п. 0000062891 00000 п. 0000065413 00000 п. 0000065491 00000 п. 0000068014 00000 п. 0000068092 00000 п. 0000068285 00000 п. 0000068363 00000 п. 0000070886 00000 п. 0000070964 00000 п. 0000073494 00000 п. 0000073572 00000 п. 0000076087 00000 п. 0000076165 00000 п. 0000078804 00000 п. 0000078882 00000 п. 0000081399 00000 п. 0000081477 00000 п. 0000083993 00000 п. 0000084071 00000 п. 0000086597 00000 п. 0000086675 00000 п. 0000089193 00000 п. 0000089271 00000 п. 00000
00000 п. 00000 00000 п. 0000094524 00000 п.
 Дети, не умеющие писать, за короткий период времени овладеют навыком правильного написания цифр.
Дети, не умеющие писать, за короткий период времени овладеют навыком правильного написания цифр.