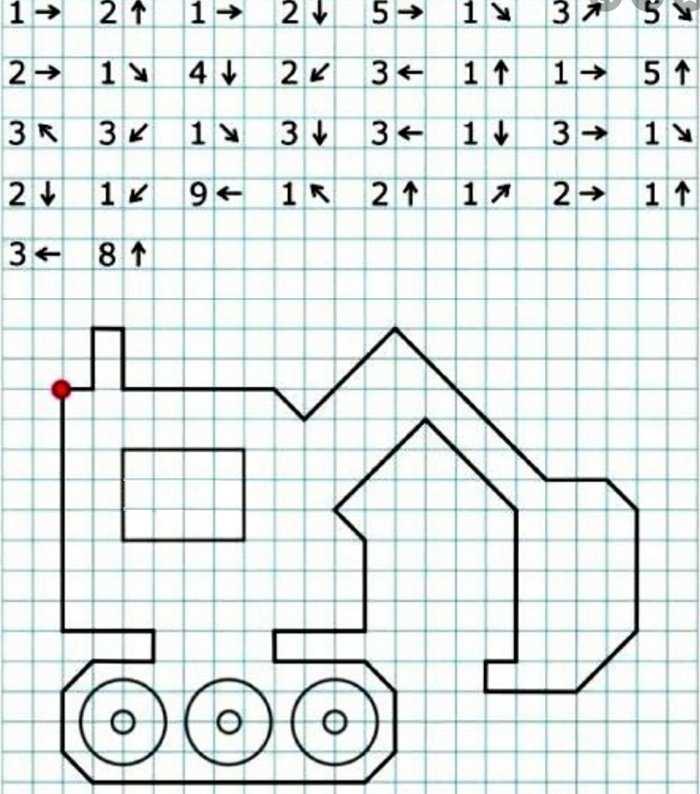
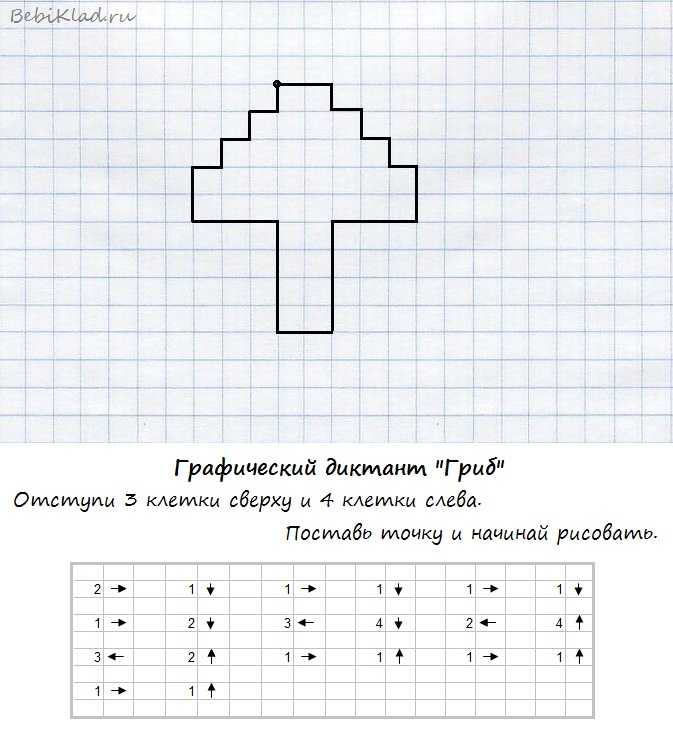
Графический диктант 1 класс по клеточкам картинки: Ничего не найдено для Graficheskij Diktant Po Kletochkam Raspechatat %23I
Графический диктант развивающие игры для детей Vector Image
Графический диктант развивающие игры для детей Vector Image- лицензионные векторы
- диктант векторов
ЛицензияПодробнее
Стандарт Вы можете использовать вектор в личных и коммерческих целях. Расширенный Вы можете использовать вектор на предметах для перепродажи и печати по требованию.Тип лицензии определяет, как вы можете использовать этот образ.
| Станд. | Расшир. | |
|---|---|---|
| Печатный/редакционный | ||
| Графический дизайн | ||
| Веб-дизайн | ||
| Социальные сети | ||
| Редактировать и изменить | ||
| Многопользовательский | ||
| Предметы перепродажи | ||
| Печать по требованию |
Владение Узнать больше
Эксклюзивный Если вы хотите купить исключительно этот вектор, отправьте художнику запрос ниже: Хотите, чтобы это векторное изображение было только у вас? Эксклюзивный выкуп обеспечивает все права этого вектора.
Мы удалим этот вектор из нашей библиотеки, а художник прекратит продажу работ.
Способы покупкиСравнить
Плата за изображение $ 14,99 Кредиты $ 1,00 Подписка 9 долларов0082 0,69Оплатить стандартные лицензии можно тремя способами. Цены составляют $ $.
| Оплата с помощью | Цена изображения |
|---|---|
| Плата за изображение $ 14,99 Одноразовый платеж | |
| Предоплаченные кредиты $ 1 Загружайте изображения по запросу (1 кредит = 1 доллар США). Минимальная покупка 30р. | |
| План подписки От 69 центов Выберите месячный план. Неиспользованные загрузки автоматически переносятся на следующий месяц. | |
Способы покупкиСравнить
Плата за изображение $ 39,99 Кредиты $ 30,00 Существует два способа оплаты расширенных лицензий.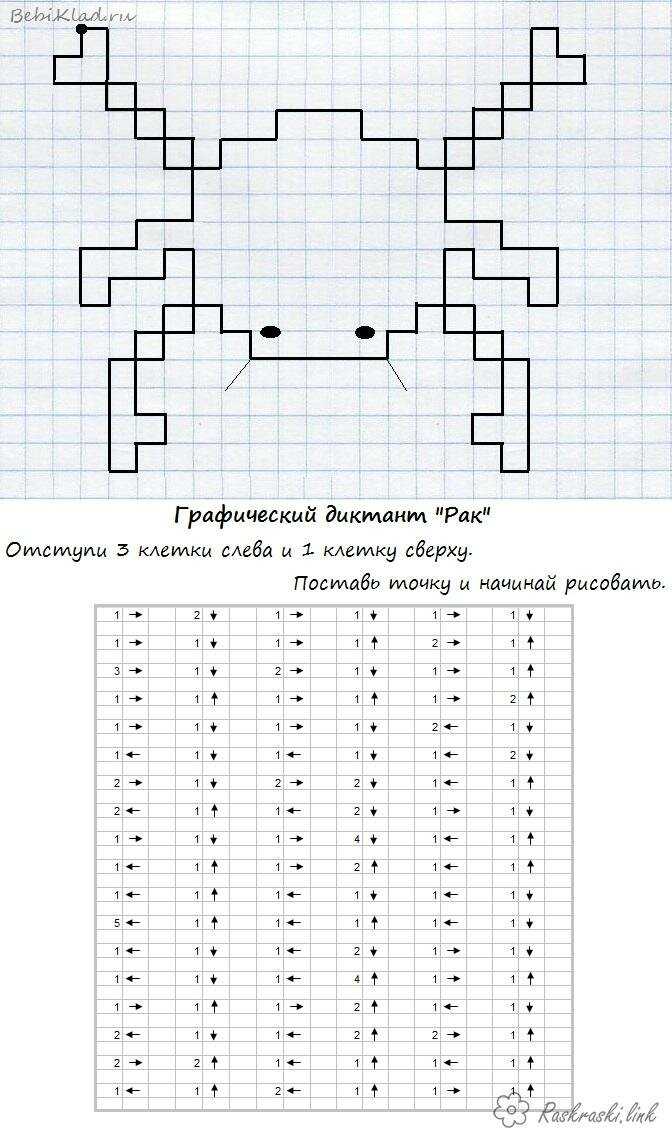
| Оплата с помощью | Стоимость изображения |
|---|---|
| Плата за изображение $ 39,99 Оплата разовая, регистрация не требуется. | |
| Предоплаченные кредиты $ 30 Загружайте изображения по запросу (1 кредит = 1 доллар США). | |
Оплата
Плата за изображение $ 499Дополнительные услугиПодробнее
Настроить изображение Доступно только с оплатой за изображение 9 долларов0082 85,00Нравится изображение, но нужно всего лишь несколько модификаций? Пусть наши талантливые художники сделают всю работу за вас!
Мы свяжем вас с дизайнером, который сможет внести изменения и отправить вам изображение в выбранном вами формате.
Примеры
- Изменить текст
- Изменить цвета
- Изменить размер до новых размеров
- Включить логотип или символ
- Добавьте название своей компании или компании
Включенные файлы
Подробности загрузки. ..
..
- Идентификатор изображения
- 42820462
- Цветовой режим
- RGB
- Художник
- юлисавко
Искусственный интеллект в радиологии – PMC
1. Редакторы Н. Благоприятное машинное обучение. Нац. Биомед. Инженер 1, 0036 (2017). [Google Scholar]
2. Мних В. и другие. Контроль на уровне человека посредством глубокого обучения с подкреплением. Природа 518, 529–533 (2015). [PubMed] [Google Scholar]
3. Моравчик М. и другие. DeepStack: искусственный интеллект экспертного уровня в безлимитном покере один на один. Наука 356, 508–513 (2017). [PubMed] [Google Scholar]
4. Сюн В. и другие. К человеческому паритету в распознавании разговорной речи. IEEE/ACM Trans. Языковой процесс аудио речи 25, 2410–2423 (2017). [Академия Google]
5. Пендлтон С.Д.
и другие.
Восприятие, планирование, контроль и координация для автономных транспортных средств. Машины
5, 6 (2017). [Google Scholar]
Машины
5, 6 (2017). [Google Scholar]
6. Серебряный D и другие. Освоение игры в го с глубокими нейронными сетями и поиском по дереву. Природа 529, 484–489 (2016). [PubMed] [Google Scholar]
7. Грейс К., Сальватье Дж., Дефо А., Чжан Б. и Эванс О. Когда ИИ превзойдет возможности человека? Доказательства от экспертов по ИИ. Препринт на arXiv, 1705.08807 (2017). [Google Scholar]
8. Раск Н. Глубокое обучение. Нац. Методы 13, 35–35 (2015). [Академия Google]
9. Эстева А. и другие. Классификация рака кожи на уровне дерматологов с глубокими нейронными сетями. Природа 542, 115–118 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
10. Гульшан В. и другие. Разработка и валидация алгоритма глубокого обучения для выявления диабетической ретинопатии на фотографиях глазного дна сетчатки. ДЖАМА 316, 2402–2410 (2016). [PubMed] [Google Scholar]
11. Аэртс HJWL
Потенциал радиометрического фенотипирования в точной медицине: обзор. JAMA Онкол. 2, 1636–1642 (2016).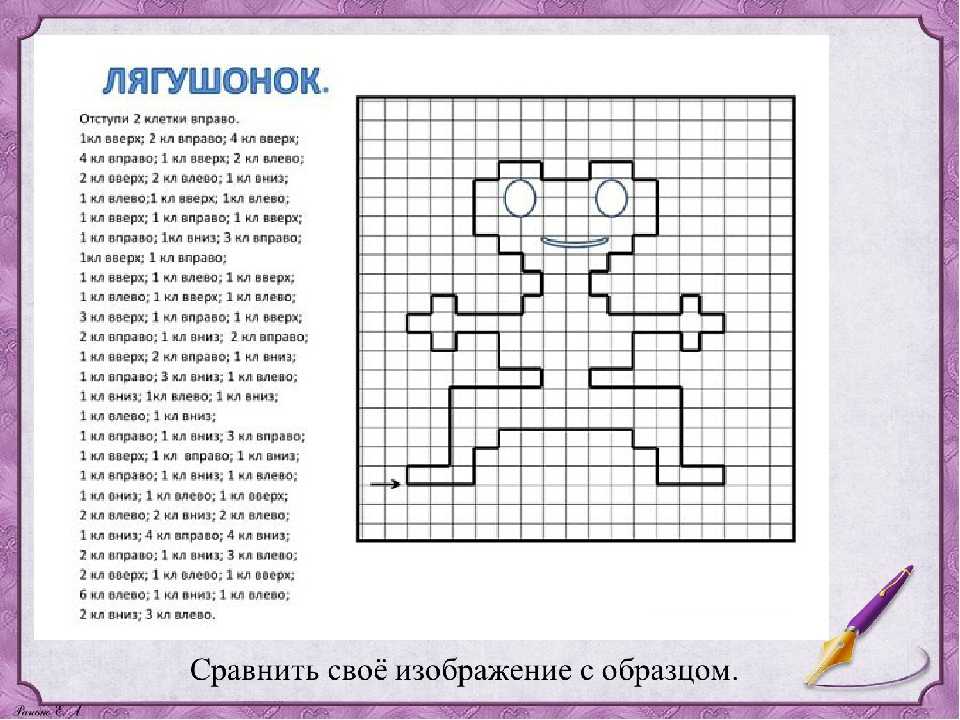 [PubMed] [Академия Google]
[PubMed] [Академия Google]
12. Кумар В. и другие. Радиомика: процесс и проблемы. Магн. Резон. Имаг 30, 1234–1248 (2012). [Бесплатная статья PMC] [PubMed] [Google Scholar]
13. Ламбин П. и другие. Радиомика: извлечение дополнительной информации из медицинских изображений с использованием расширенного анализа признаков. Евро. Дж. Рак 48, 441–446 (2012). [Бесплатная статья PMC] [PubMed] [Google Scholar]
14. Litjens G и другие. Обзор по глубокому обучению в анализе медицинских изображений. Мед. Анальный образ 42, 60–88 (2017). [PubMed] [Академия Google]
15. Колоссвари М., Келлермайер М., Меркели Б. и Маурович-Хорват П. Радиомика компьютерной томографии сердца: всесторонний обзор радиомикологических методов. Дж. Торак. Имаг 33, 26–34 (2018). [PubMed] [Google Scholar]
16. Аэртс HJWL и другие. Расшифровка фенотипа опухоли с помощью неинвазивной визуализации с использованием количественного радиомического подхода. Нац. Сообщество 5, 4006 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
17. Coroller TP
и другие.
Рентгенологическая сигнатура на основе КТ предсказывает отдаленные метастазы аденокарциномы легкого. Радиотер. Онкол
114, 345–350 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Coroller TP
и другие.
Рентгенологическая сигнатура на основе КТ предсказывает отдаленные метастазы аденокарциномы легкого. Радиотер. Онкол
114, 345–350 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
18. Ву В и другие. Поисковое исследование для определения классификаторов радиомики для гистологии рака легких. Передний. Онкол 6, 71 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
19. Huynh E и другие. Ассоциации рентгенологических данных, извлеченных из статических и дыхательных КТ-сканирований, с рецидивом заболевания у пациентов с раком легких, получавших SBRT. ПЛОС ОДИН 12, e0169172 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
20. Rios Velazquez E и другие. Соматические мутации определяют различные фенотипы визуализации при раке легкого. Рак рез. 77, 3922–3930 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
21. Grossmann P и другие. Определение биологической основы радиомного фенотипа при раке легкого. электронная жизнь 6, e23421 (2017) [бесплатная статья PMC] [PubMed] [Google Scholar]
22.
23. O’Connor JPB и другие. Дорожная карта биомаркеров изображений для исследований рака. Нац. Преподобный Клин. Онкол 14, 169–186 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
24. Boland GWL, Guimaraes AS & Mueller PR Головоломка рентгенолога: преимущества и издержки увеличения мощности и использования КТ. Евро. Радиол 19, 9–12 (2009). [PubMed] [Google Scholar]
25. McDonald RJ и другие. Влияние изменений в использовании и технологических достижений визуализации поперечного сечения на рабочую нагрузку рентгенолога. акад. Радиол 22, 1191–1198 (2015). [PubMed] [Google Scholar]
26. Фицджеральд Р. Ошибка в рентгенологии. клин. Радиол 56, 938–946 (2001). [PubMed] [Google Scholar]
27. Ledley RS & Lusted LB
Аргументационные основы медицинской диагностики; символическая логика, теория вероятности и ценности помогают нам понять, как рассуждают врачи.
28. Лодвик Г.С., Китс Т.Е. и Дорст Дж.П. Кодирование рентгенограмм для компьютерного анализа применительно к раку легкого. Радиология 81, 185–200 (1963). [PubMed] [Google Scholar]
29. Ambinder EP История перехода к полной компьютеризации медицины. Дж. Онкол. Практика 1, 54–56 (2005). [Бесплатная статья PMC] [PubMed] [Google Scholar]
30. Хауг П.Дж. Использование диагностических экспертных систем в клинической практике. проц. Анну. Симп. вычисл. заявл. Мед. Уход, 379–383 (1993). [Бесплатная статья PMC] [PubMed] [Google Scholar]
31. Кастеллино Р.А. Компьютерное обнаружение (CAD): обзор. Образ Рака. 5, 17–19 (2005). [Бесплатная статья PMC] [PubMed] [Google Scholar]
32. Shen D, Wu G & Suk H-I Глубокое обучение в анализе медицинских изображений. Анну. Преподобный Биомед. анг 19, 221–248 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
33. Виарагаван Х
MO-A-207B-01: Радиомика: методы сегментации и извлечения признаков. Мед. Физ.
43, 3694–3694 (2016). [Google Scholar]
Мед. Физ.
43, 3694–3694 (2016). [Google Scholar]
34. Пол Р. и другие. Глубокое обучение переносу признаков в сочетании с традиционными признаками предсказывает выживаемость пациентов с аденокарциномой легкого. Томография 2, 388–395 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
35. Cheng J-Z и другие. Компьютерная диагностика с архитектурой глубокого обучения: применение к поражениям молочной железы на изображениях УЗИ и узлам в легких на КТ. науч. представитель 6, 24454 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
36. Chen H, Zheng Y, Park J-H, Heng P-A & Zhou SK в области обработки медицинских изображений и компьютерных вмешательств — MICCAI 2016 487–495 (Афины, Греция, 2016 г.). [Google Scholar]
37. Гафурян М. и другие. Глубокие сверточные нейронные сети, чувствительные к местоположению, для сегментации гиперинтенсивности белого вещества. науч. представитель 7, 5110 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
38. Ван Х.
и другие.
Сравнение методов машинного обучения для классификации метастазов немелкоклеточного рака легкого в лимфатические узлы средостения по изображениям ПЭТ/КТ с 18F-ФДГ. EJNMMI Рез. 7, 11 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Ван Х.
и другие.
Сравнение методов машинного обучения для классификации метастазов немелкоклеточного рака легкого в лимфатические узлы средостения по изображениям ПЭТ/КТ с 18F-ФДГ. EJNMMI Рез. 7, 11 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
39. Ван Гиннекен Б., Шефер-Прокоп К.М. и Прокоп М. Компьютерная диагностика: как перейти из лаборатории в клинику. Радиология 261, 719–732 (2011). [PubMed] [Google Scholar]
40. Нагарадж С., Рао Г.Н. и Котешварарао К. Роль распознавания образов в компьютерной диагностике и компьютерного обнаружения в медицинской визуализации: клиническая проверка. Междунар. Дж. Вычисл. Приложение 8, 18–22 (2010). [Google Scholar]
41. Коул Э.Б. и другие. Влияние компьютерных систем обнаружения на точность рентгенолога при цифровой маммографии. AJR Ам. Дж. Рентгенол 203, 909–916 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
42. Компакт-диск Lehman
и другие.
Диагностическая точность цифровой скрининговой маммографии с компьютерным обнаружением и без него. JAMA Стажер. Мед
175, 1828–1837 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
JAMA Стажер. Мед
175, 1828–1837 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
43. Huang X, Shan J & Vaidya V в 2017 г. IEEE 14 th International Symposium on Biomedical Imaging (ISBI 2017) 379–383 (Мельбурн, Австралия, 2017 г.). [Google Scholar]
44. Цехай Ю.К. и другие. в трудах SPIE https://doi.org/10.1117/12.2254423 (2017 г.).
45. Кои Т. и другие. Крупномасштабное глубокое обучение для компьютерного обнаружения маммографических поражений. Мед. Анальный образ 35, 303–312 (2017). [PubMed] [Google Scholar]
46. Шарма Н. и Аггарвал Л.М. Методы автоматизированной сегментации медицинских изображений. Дж. Мед. Физ. 35, 3–14 (2010). [Бесплатная статья PMC] [PubMed] [Google Scholar]
47. Харалик Р.М. и Шапиро Л.Г. Методы сегментации изображения. График компьютерного зрения. Процесс изображения 29, 100–132 (1985). [Google Scholar]
48. Фам Д.Л., Сюй С. и Принц Дж.Л.
Современные методы сегментации медицинских изображений. Анну. Преподобный Биомед. анг
2, 315–337 (2000). [PubMed] [Академия Google]
Анну. Преподобный Биомед. анг
2, 315–337 (2000). [PubMed] [Академия Google]
49. Грау В., Мьюс А.Ю., Альканьис М., Кикинис Р. и Уорфилд СК Улучшенное преобразование водораздела для сегментации медицинских изображений с использованием априорной информации. IEEE транс. Мед. Имаг 23, 447–458 (2004). [PubMed] [Google Scholar]
50. Паризо С. и другие. Вероятностный атлас локализации диффузных глиом II степени ВОЗ в головном мозге. ПЛОС ОДИН 11, e0144200 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
51. Ghose S и другие. в 2012 г. 19-я Международная конференция IEEE по обработке изображений. 541–544 (Орландо, Флорида, США, 2012 г.). [Академия Google]
52. Хан Х и другие. Автоматическая сегментация КТ головы и шеи на основе атласа. Мед. Вычисление изображения. вычисл. Ассистент Интерв 11, 434–441 (2008). [PubMed] [Google Scholar]
53. Лонг Дж., Шелхамер Э. и Даррелл Т. на конференции IEEE 2015 г. по компьютерному зрению и распознаванию образов (CVPR)
3431–3440 (Бостон, Массачусетс, США, 2015 г. ). [Google Scholar]
). [Google Scholar]
54. Ronneberger O, Fischer P & Brox TU in Medical Image Computing and Computer-Assisted Intervention — MICCAI 2015 234–241 (Мюнхен, Германия, 2015 г.). [Академия Google]
55. Моескопс П и другие. в области вычислений медицинских изображений и компьютерных вмешательств – MICCAI 2016 478–486 (Афины, Греция, 2016 г.). [Google Scholar]
56. Де Бребиссон А. и Монтана Г. в 2015 г. Конференция IEEE по компьютерному зрению и семинарам по распознаванию образов (CVPRW) 20–28 (Бостон, Массачусетс, США, 2015 г.). [Google Scholar]
57. Чоффи У., Равелья Ф., Де Симоне М. и Байси А. Помутнение по типу матового стекла: излечимое заболевание, но большая проблема для хирургов. Дж. Торак. Кардиовас. Сур 154, 375–376 (2017). [PubMed] [Академия Google]
58. Шампейн Д.Л. и Седербом Г.Дж. Достижения в обнаружении рака молочной железы с помощью скрининговой маммографии. Окснер Дж. 2, 33–35 (2000). [Бесплатная статья PMC] [PubMed] [Google Scholar]
59. Shiraishi J, Li Q, Appelbaum D & Doi K
Компьютерная диагностика и искусственный интеллект в клинической визуализации. Семин. Нукл. Мед
41, 449–462 (2011). [PubMed] [Google Scholar]
Shiraishi J, Li Q, Appelbaum D & Doi K
Компьютерная диагностика и искусственный интеллект в клинической визуализации. Семин. Нукл. Мед
41, 449–462 (2011). [PubMed] [Google Scholar]
60. Айер Т., Айвачи М.Ю., Лю З.С., Алагоз О. и Бернсайд Э.С. Компьютерные диагностические модели в скрининге рака молочной железы. Изображение Мед 2, 313–323 (2010). [Бесплатная статья PMC] [PubMed] [Google Scholar]
61. Чжан Дж, Ван Ю, Ю Б, Ши Х и Чжан Ю Применение компьютерной диагностики для ультразвуковой оценки шейных лимфатических узлов. Ультрасон. Имаг 38, 159–171 (2016). [PubMed] [Google Scholar]
62. Джаннини В. и другие. Полностью автоматическая система компьютерной диагностики рака предстательной железы периферической зоны с помощью мультипараметрической магнитно-резонансной томографии. вычисл. Мед. График визуализации 46, 219–226 (2015). [PubMed] [Google Scholar]
63. Эль-Баз А.
и другие.
Системы компьютерной диагностики рака легкого: проблемы и методологии. Междунар. Дж. Биомед. Имаг
2013, 942353 (2013). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Дж. Биомед. Имаг
2013, 942353 (2013). [Бесплатная статья PMC] [PubMed] [Google Scholar]
64. Edey AJ & Hansell DM Случайно обнаружены мелкие легочные узелки на КТ. клин. Радиол 64, 872–884 (2009). [PubMed] [Google Scholar]
65. Mirsadraee S, Oswal D, Alizadeh Y, Caulo A & van Beek E Jr. 7-я система классификации и стадирования рака легкого по TNM: обзор изменений и последствий. Мир Дж. Радиол 4, 128–134 (2012). [Бесплатная статья PMC] [PubMed] [Google Scholar]
66. Sohn K, Shang W & Lee H в Advances in Neural Information Processing Systems 27 (NIPS 2014) (ред. Ghahramani Z, Welling M, Cortes C, Lawrence ND & Вайнбергер KQ) 2141–2149(Монреаль, Канада, 2014 г.). [Google Scholar]
67. Литьенс Г. и другие. Глубокое обучение как инструмент повышения точности и эффективности гистопатологической диагностики. науч. представитель 6, 26286 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
68. Круз-Роа А.
и другие.
Точное и воспроизводимое обнаружение инвазивного рака молочной железы на изображениях всего предметного стекла: подход глубокого обучения для количественной оценки распространенности опухоли. науч. представитель
7, 46450 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
науч. представитель
7, 46450 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
69. Джефф CC Меры реагирования: RECIST, ВОЗ и новые альтернативы. Дж. Клин. Онкол 24, 3245–3251 (2006). [PubMed] [Google Scholar]
70. Тисс П. и другие. Точность частоты ответов в онкологических исследованиях: причины изменчивости между наблюдателями. Groupe Français d’Immunothérapie Национальной федерации центров борьбы с раком. Дж. Клин. Онкол 15, 3507–3514 (1997). [PubMed] [Google Scholar]
71. Хорасани Р., Эриксон Б.Дж. и Патриарх Дж. Новые возможности компьютерной диагностики: обнаружение изменений и характеристика. Варенье. Сб. Радиол 3, 468–469(2006). [PubMed] [Google Scholar]
72. Патриарх Дж. В. и Эриксон Б. Дж. Часть 1. Автоматическое обнаружение и характеристика изменений в серийных МРТ-исследованиях пациентов с опухолью головного мозга. Дж. Цифра. Имаг 20, 203–222 (2007). [Бесплатная статья PMC] [PubMed] [Google Scholar]
73. Pan X, Sidky EY & Vannier M
Почему коммерческие компьютерные томографы до сих пор используют традиционную фильтрованную обратную проекцию для реконструкции изображения?
Обратная задача
25, 1230009 (2009). [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
74. Пипатрисават Т., Гачич А., Франкетти Ф., Пушель М. и Моура Дж.М.Ф. в материалах дела. (ИКАССП ‘05). Международная конференция IEEE по акустике, речи и обработке сигналов, 2005 г. v/153–v/156 (Филадельфия, Пенсильвания, США, 2005 г.). [Google Scholar]
75. Чжу Б., Лю Дж. З., Коли С. Ф., Розен Б. Р. и Розен М. С. Реконструкция изображения с помощью обучения множеству доменных преобразований. Природа 555, 487–492 (2018). [PubMed] [Google Scholar]
76. Hammernik K, Würfl T, Pock T & Maier AA in Bildverarbeitung für die Medizin 2017 (eds Maier-Hein K, Deserno T, Handels H & Tolxdorff T) 92–97 (Спрингер, Берлин, Гейдельберг, 2017). [Google Scholar]
77. Гьестеби Л. и другие. in Разработки в области рентгеновской томографии XI 10391–31 (Сан-Диего, Калифорния, США, 2017 г.). [Google Scholar]
78. Эль-Гамаль ФЭ-ЗА, Эльмоги М. и Атван А.
Современные тенденции в регистрации и слиянии медицинских изображений. Египет. информ. Дж
17, 99–124 (2016). [Google Scholar]
Египет. информ. Дж
17, 99–124 (2016). [Google Scholar]
79. Yang X, Kwitt R, Styner M & Niethammer M Quicksilver: быстрая предиктивная регистрация изображений — подход к глубокому обучению. Нейроизображение 158, 378–396 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
80. Нгиам Дж. и другие. в материалах 28-й Международной конференции по машинному обучению 689–696 (Белвью, Вашингтон, США, 2011 г.). [Google Scholar]
81. Янкеелов Т.Е., Абрамсон Р.Г., Куорлз С.К. Количественная мультимодальная визуализация в исследованиях и терапии рака. Нац. Преподобный Клин. Онкол 11, 670–680 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
82. Джонсон А.Дж., Чен М.М., Западка М.Е., Лайдерс Э.М. и Литтенберг Б. Ясность радиологического отчета: когортное исследование структурированного отчета по сравнению с обычной диктовкой. Варенье. Сб. Радиол 7, 501–506 (2010). [PubMed] [Академия Google]
83. Леви М.А. и Рубин Д.Л.
Инструментальная поддержка для оценки клинического ответа на лечение. АМИЯ Анну. Симп. Проц
2008, 399–403 (2008). [Бесплатная статья PMC] [PubMed] [Google Scholar]
АМИЯ Анну. Симп. Проц
2008, 399–403 (2008). [Бесплатная статья PMC] [PubMed] [Google Scholar]
84. Европейское общество радиологии (ESR). Надлежащая практика радиологической отчетности. Рекомендации Европейского общества радиологии (ESR). Инсайты 2, 93–96 (2011). [Бесплатная статья PMC] [PubMed] [Google Scholar]
85. Folio LR и другие. Количественная радиологическая отчетность в онкологии: опрос онкологов и рентгенологов. AJR Ам. Дж. Рентгенол 205, W233–W243 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
86. Карпаты А. и Фей-Фей Л. Глубокое визуально-семантическое выравнивание для генерации описаний изображений. IEEE транс. Анальный узор. Мах. Интелл 39, 664–676 (2017). [PubMed] [Google Scholar]
87. Shin HC и другие. в 2016 г. на конференции IEEE по компьютерному зрению и распознаванию образов (CVPR) 2497–2506 (Лас-Вегас, Невада, США, 2016 г.). [Google Scholar]
88. Ли Дж. Г.
и другие.
Глубокое обучение в медицинской визуализации: общий обзор. Кор. Дж. Радиол
18, 570–584 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Кор. Дж. Радиол
18, 570–584 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
89. ОЭСР. Компьютерная томография (КТ). https://doi.org/10.1787/3c994537-en (2018 г.).
90. ОЭСР. Магнитно-резонансная томография (МРТ). https://doi.org/10.1787/1d89353f-en (2018 г.).
91. Брайан С. и другие. Время отчетов радиологии: влияние систем архивации изображений и связи. AJR Ам. Дж. Рентгенол 170, 1153–1159 (1998) [PubMed] [Google Scholar]
92. Мансури Б., Эрхард К.К. и Саншайн Дж.Л. Внедрение системы архивирования и передачи изображений (PACS), ее интеграция и преимущества в интегрированной системе здравоохранения. акад. Радиол 19, 229–235 (2012). [PubMed] [Google Scholar]
93. Лемке Х.У. Развитие PACS в Европе. вычисл. Мед. Изображение График 27, 111–120 (2003). [PubMed] [Google Scholar]
94. Мендель Дж. Б. и Швейцер А. Л. PACS для развивающихся стран. Дж. Глобал Радиол 1, 5 (2015). [Google Scholar]
95. Гудфеллоу I
и другие. в Advances in Neural Information Processing Systems 27 (NIPS 2014) (редакторы Ghahramani Z, Welling M, Cortes C, Lawrence ND & Weinberger KQ) 2672–2680 (Монреаль, Канада, 2014). [Академия Google]
[Академия Google]
96. Kingma DP и Welling M Автокодирование вариационного Байеса. Препринт на arXiv, 1312.6114 (2013). [Google Scholar]
97. Камницас К. и другие. в обработке информации в медицинской визуализации 597–609 (Спрингер, Чам, 2017). [Google Scholar]
98. Калленберг М. и другие. Неконтролируемое глубокое обучение применяется к сегментации плотности груди и оценке маммографического риска. IEEE транс. Мед. Имаг 35, 1322–1331 (2016). [PubMed] [Google Scholar]
99. Чжан П., Ван Ф и Чжэн И в 2017 г. IEEE 14 -й Международный симпозиум по биомедицинской визуализации (ISBI 2017) 578–582 (Мельбурн, Австралия, 2017 г.). [Google Scholar]
100. Кларк К. и другие. Архив изображений рака (TCIA): поддержание и эксплуатация общедоступного хранилища информации. Дж. Цифра. Имаг 26, 1045–1057 (2013). [Бесплатная статья PMC] [PubMed] [Google Scholar]
101. Ван Г.А. Перспектива глубокой визуализации. IEEE-доступ 4, 8914–8924 (2016). [Google Scholar]
102. Ford RA, Price W & Nicholson II
Конфиденциальность и подотчетность в медицине черного ящика. Мичиган Телекоммуникации. Тех. Л. Преподобный
23, 1 (2016). [Академия Google]
Ford RA, Price W & Nicholson II
Конфиденциальность и подотчетность в медицине черного ящика. Мичиган Телекоммуникации. Тех. Л. Преподобный
23, 1 (2016). [Академия Google]
103. Селбст А.Д. и Паулз Дж. Значимая информация и право на объяснение. Междунар. Закон о конфиденциальности данных 7, 233–242 (2017). [Google Scholar]
104. Имминг П., Синнинг С. и Мейер А. Наркотики, их мишени, характер и количество мишеней. Нац. Преподобный Друг Дисков 5, 821–834 (2006). [PubMed] [Google Scholar]
105. Мельхорн Х. и другие. в Энциклопедии паразитологии 3-е изд. (изд. Mehlhorn H) 400–402 (Springer, Berlin, Heidelberg, 2008). [Google Scholar]
106. Шокри Р, Шматиков В in Proceedings of the 22 nd Конференция ACM SIGSAC по компьютерной и коммуникационной безопасности 1310–1321 (Денвер, Колорадо, США, 2015 г.). [Google Scholar]
107. Фонг Л.Т., Аоно Ю., Хаяши Т., Ван Л. и Мориай С. Приложения и методы информационной безопасности. 8-я международная конференция, ATIS 2017 (ред. Баттен Л., Ким Д.С., Чжан С. и Ли Г.) 719, 100–110 (Окленд, Новая Зеландия, 2017). [Google Scholar]
Баттен Л., Ким Д.С., Чжан С. и Ли Г.) 719, 100–110 (Окленд, Новая Зеландия, 2017). [Google Scholar]
108. McMahan HB, Moore E, Ramage D, Hampson S & y Arcas BA в материалах 20-й Международной конференции по искусственному интеллекту и статистике (AISTATS) 1273–1282 (Форт-Лодердейл, Флорида, США, 2017 г.). [Академия Google]
109. Гилад-Бахрах Р. и другие. в материалах 33-й -й -й Международной конференции по машинному обучению 201–210 (Нью-Йорк, США, 2016 г.). [Google Scholar]
110. Кахан А. и Чимино Дж.Дж. Обучение системы здравоохранения с помощью компьютерной диагностики. Дж. Мед. Интернет Рез 19, e54 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
111. LeCun Y, Bengio Y & Hinton G Глубокое обучение. Природа 521, 436–444 (2015). [PubMed] [Google Scholar]
112. Miotto R, Wang F, Wang S, Jiang X & Dudley JT
Глубокое обучение для здравоохранения: обзор, возможности и проблемы. Краткий. Биоинформ
https://doi.org/10.1093/биб/bbx044 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
113. Кевин Чжоу С., Гринспен Х. и Шен Д. Глубокое обучение для анализа медицинских изображений. (Академическая пресса, 2017). [Google Scholar]
114. Шин Х-С и другие. Глубокие сверточные нейронные сети для компьютерного обнаружения: архитектуры CNN, характеристики набора данных и трансферное обучение. IEEE транс. Мед. Имаг 35, 1285–1298 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
115. Shin Y & Balasingham I в 2017 г. 39Ежегодная международная конференция IEEE Engineering in Medicine and Biology Society (EMBC) 3277–3280 (остров Чеджудо, Корея, 2017 г.). [Google Scholar]
116. Оррингер Д.А. и другие. Быстрая интраоперационная гистология необработанных хирургических образцов с помощью микроскопии стимулированного комбинационного рассеяния на основе волоконного лазера. Нац. Биомед. анг 1, 0027 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
117. Альбаркуни С.
и другие.
AggNet: глубокое изучение толпы для обнаружения митоза на гистологических изображениях рака молочной железы.